В 2025 году журналу
ИСПОЛНИТСЯ
95 ЛЕТ!
ЧИТАЙТЕ
Материалы к юбилею >>>

РУБРИКИ
 Племенное дело |
 Крупный план |
 Актуально |
 Корма |
 Техническая политика |
 Событие |
 Ветеринария |
 Эксклюзивное интервью |
 Статистика |
 Выставки |
Творчество
наших читателей
ЧИТАЙТЕ
Автор С.И. КАЛИНИЧЕНКО
ГИМН СВИНЬЕ >>>
СТАТЬИ ИЗ НОМЕРА
DOI: 10.37925/0039-713X-2024-8-55-60
УДК 636.02/636.03/636.4.033
ГЕНЕТИЧЕСКАЯ ИЗОЛЯЦИЯ И ИДЕНТИЧНОСТЬ СВИНЕЙ РОССИЙСКОЙ И ЗАРУБЕЖНОЙ ПОПУЛЯЦИИ
А.А. БЕЛОУС, кандидат биолог. наук, ст. научный сотрудник, e-mail: belousa663@gmail.com, А.С. АБДЕЛЬМАНОВА, доктор биолог. наук, ст. научный сотрудник, В.Р. ХАРЗИНОВА, кандидат биолог. наук, вед. научный сотрудник, Н.А. ЗИНОВЬЕВА, доктор биолог. наук, профессор, академик РАН, директор, ФГБНУ ФИЦ ВИЖ имени Л.К. Эрнста
Понимание генетического однообразия пород сельскохозяйственных животных необходимо для установления исторических аспектов их формирования. В связи с чем были исследованы три контрастные породы – локальная российская ливенская, китайская сутай и крупная белая порода свиней. Проведен расчет генетической дифференциации статистическим методом FST пород попарно друг с другом, выявлены функционально значимые гены и проанализирована их биологическая аннотация.
Ключевые слова: FST, свиньи породы крупная белая, свиньи ливенской породы, свиньи китайской породы сутай.
Genetic isolation and identity in pigs of Russian and foreign populations
A.A. BELOUS, candidate of biological sciences, senior researcher, e-mail: belousa663@gmail.com, A.S. ABDELMANOVA, doctor of biological sciences, senior researcher, V.R. KHARZINOVA, candidate of biological sciences, leading researcher, N.A. ZINOVYEVA, doctor of biological sciences, professor, academician of the RAS, director, Federal Research Center for Animal Husbandry named after academy member L.K. Ernst
Understanding the genetic uniformity of farm animal breeds is necessary to establish the historical aspects of their formation. In this regard, we took three contrasting breeds – local Russian Liven breed, Chinese Sutai breed and Large White pig breed. The calculation of genetic differentiation by the statistical method FST breeds in pairs with each other was carried out, functionally significant genes were identified and their biological annotation was analyzed.
Key words: FST, Large White pigs, Livonia pigs, Chinese Sutai pigs.
Введение
В настоящее время переход к высокопродуктивному и экологически чистому агро- и аквахозяйству, создание безопасных и качественных, в том числе функциональных, продуктов питания является одним из приоритетов научно-технологического развития нашей страны [1]. Для его выполнения необходимо внедрять в системы определения и породоспецифичности животных новые генетические методы, показавшие достоверность при их применении.
За последние 30 лет было приложено много усилий исследователей для характеристики пород сельскохозяйственных животных с помощью молекулярной генетики, в первую очередь посредством технологий микросателлитов (MS) и однонуклеотидного полиморфизма (SNP). Данные исследования обычно направлены на поддержку сохранения и устойчивого использования биоразнообразия животных, а также на выяснение процессов одомашнивания, эволюции и дифференциации пород.
Одним из результатов таких исследований является расчет степени, в которой породы расходятся друг с другом. Широко используемой мерой для получения генетической дифференциации стала статистика FST. Как первоначально было описано, значения FST от 0,05 до 0,15 приняты для указания на умеренную дифференциацию между популяциями, от 0,15 до 0,25 – на высокую дифференциацию, а больше 0,25 – на очень высокую дифференциацию [2]. В принципе FST можно было бы применять для информирования обсуждений, касающихся конкретных пород, например, что они достаточно отличаются друг от друга, чтобы оправдать поддержку их сохранения, или наоборот, что они весьма похожи, чтобы их можно было объединить.
В практическом направлении измерения FST нечасто используются в качестве основного генетического обоснования для принятия решений по сохранению пород, но большое количество доступных измерений FST представляет собой ресурс данных, который может дать представление об общих закономерностях дифференциации пород. Действительно, генетическая дифференциация пород нередко помещается в пространственный контекст путем исследования того, как она соотносится с географическим расстоянием [3]. Некоторые исследования также показали корреляцию с человеческим [4, 5] и экологическим разнообразием [6].
Целью исследования явилось изучение генетического однообразия между китайской породой свиней сутай, коммерческой породой – крупной белой и уникальной локальной породой свиней в России – ливенской.
Материалы и методы исследований
Работа была выполнена в лаборатории генетических технологий в агро- и аквахозяйстве ФГБНУ ФИЦ ВИЖ имени Л.К. Эрнста с применением инвентаря Центра коллективного пользования научным оборудованием «Биоресурсы и биоинженерия сельскохозяйственных животных» ФГБНУ «Федеральный исследовательский центр животноводства – ВИЖ имени академика Л.К. Эрнста».
В исследованиях были использованы образцы (фрагмент уха) из коллекции Центра по породам свиней ливенская (n=192) и крупная белая (n=797). Выделение ДНК проводили с помощью наборов для выделения геномной ДНК серии «ДНК-Экстран» (ЗАО «Синтол», Россия) в соответствии с протоколом фирмы-производителя. Концентрацию двухцепочечной ДНК определяли посредством флуориметра Qubit 2.0 (Invitrogen/Life Technologies, США). Для определения качества ДНК измеряли соотношение OD260/OD280 (cпектрофотометр NanoDrop 8000, Thermo Fisher Scientific, США). Для анализа использовали ДНК с OD260/OD280=1,6–1,8. Кроме того, качество ДНК оценивали с помощью гель-электрофореза в 1%-ном агарозном геле. Полногеномное генотипирование производили с применением высокоплотных ДНК-чипов PorcineHD Genotyping BeadChip (платформа GeneSeek Genomic Profiler, Neogene, США), содержащим 66 763 SNP.
Файлы, содержащие геномную информацию (PED/MAP) по свиньям китайских пород, были взяты с сайта Dryad (В данном исследовании анализировали породу сутай.
Контроль качества данных, выполненный с использованием PLINK 1.90, определил удаление образцов и SNP ниже уровня генотипирования 95%.
Генетическая дифференциация, основанная на попарных значениях FST, была рассчитана между всеми SNP с использованием PLINK 1.9 и SNP, входящих в 0,1% максимальных значений FST, представлявших отпечатки селекции.
Для поиска генов-кандидатов, в области которых идентифицированы SNP, использовали геномный ресурс Sscrofa11.1 (https://www.ncbi.nlm.nih.gov/assembly/GCF_000003025.6) и Ensembl (www.ensembl.org). Функциональные аннотации генов выполняли с привлечением веб-базы DAVID (https://david.ncifcrf.gov).
Результаты исследований
После контроля качества осталось:
- по породе крупная белая – 52 484 SNP;
- по ливенской породе свиней – 56 102 SNP;
- по породе сутай – 39 695 SNP.
Среднее значение по FST группы пород крупная белая vs сутай составило 0,187, по ливенской vs сутай – 0,154 и по крупная белая vs ливенская – 0,070. Исходя из приведенных данных, наименьшая генетическая сходимость наблюдается при сравнении локальной породы России – ливенской с одной из распространенных коммерческих пород – крупной белой (рис.).
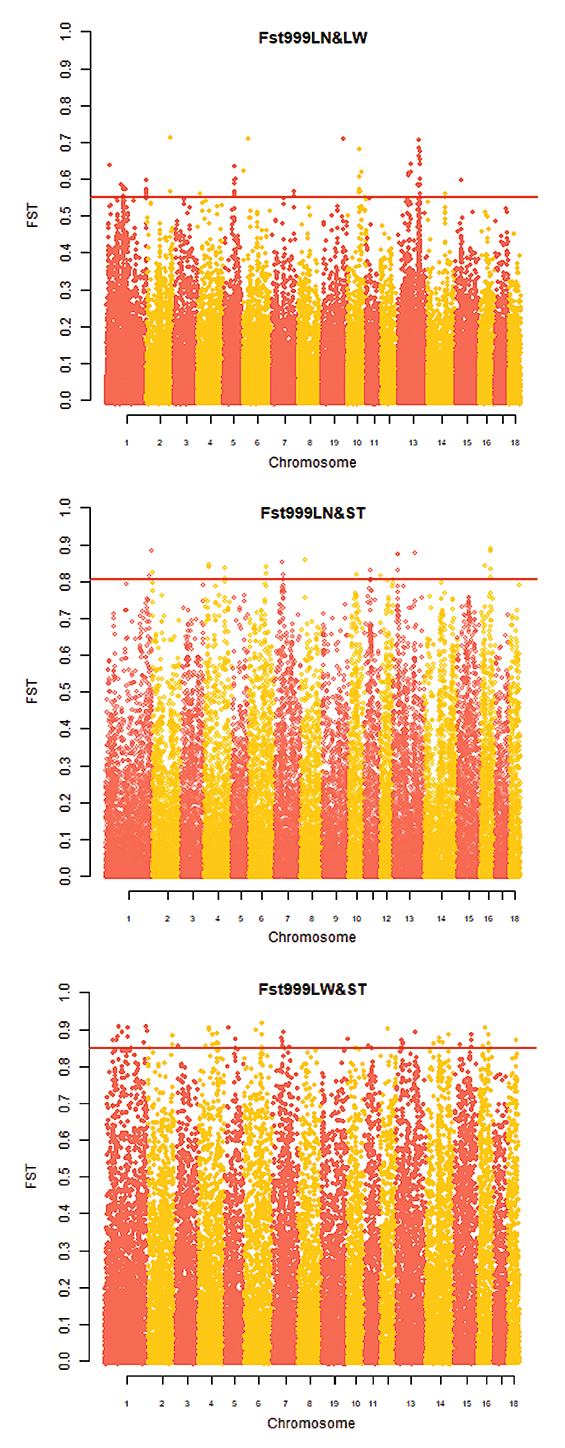
Рис. Генетическая дифференциация по значениям FST изучаемых пород свиней (сверху вниз): крупная белая vs ливенская, ливенская vs сутай и крупная белая vs сутай
Из 221 выявленного гена значимую функциональную аннотацию имели 49 генов, распределенных следующим образом: 6 генов по группе пород свиней крупная белая vs ливенская, 26 генов – по группе крупная белая vs сутай и 18 – по ливенская vs сутай (табл.).
Таблица. Функциональная аннотация
|
№ хр. |
SNPs |
Ген |
Аннотация по DAVID |
Порода |
|
1 |
ASGA0003780 |
ACAA2 |
Бета-окисление жирных кислот, клеточный ответ на гипоксию |
Крупная Белая vs ливенская |
|
ASGA0003868 |
STARD6 |
Связывание липидов |
Крупная Белая vs ливенская |
|
|
MARC0059892 |
BVES |
Развитие сердца, ткани скелетных мышц |
Крупная Белая vs ливенская |
|
|
ASGA0004750, ASGA0004753 |
CDH20 |
Развитие многоклеточного организма |
Крупная Белая vs ливенская |
|
|
ALGA0003302 |
GABRR1 |
Процесс нервной системе |
Крупная Белая vs ливенская |
|
|
ALGA0003302 |
GABRR2 |
Процесс нервной системы |
Крупная Белая vs ливенская |
|
|
MARC0088139 |
L3MBTL3 |
Созревание эритроцитов |
Крупная Белая vs ливенская |
|
|
MARC0059892 |
POPDC3 |
Развитие сердца, ткани скелетных мышц |
Крупная Белая vs ливенская |
|
|
ALGA0003302 |
UBE2J1 |
Развитие сперматид |
Крупная Белая vs ливенская |
|
|
2 |
ALGA0116847 |
CDX1 |
Развитие эмбриона, морфогенез органов животных, костей |
Крупная Белая vs ливенская |
|
MARC0066239, ALGA0104042 |
CEND1 |
Развитие мозжечка |
Ливенская vs cуйтай |
|
|
MARC0066239, ALGA0104042 |
CHID1 |
Процесс метаболизма углеводов |
Ливенскаяvscуйтай |
|
|
MARC0066239, ALGA0104042 |
TALDO1 |
Процесс углеводного обмена |
Ливенскаяvscуйтай |
|
|
4 |
ALGA0026066, ALGA0026071 |
CD247 |
Адаптивный иммунный ответ |
Крупная Белая vs ливенская |
|
ALGA0027907 |
DBT |
Процесс обмена карбоновых кислот |
Крупная Белая vs ливенская |
|
|
ALGA0027373 |
SYPL2 |
Развитие сердца |
Ливенскаяvscуйтай |
|
|
5 |
ALGA0105397, ALGA0121100, MARC0032012 |
TEAD4 |
Развитие эмбриональных органов |
Крупная Белая vs ливенская |
|
M1GA0007600 |
CACNG2 |
Развитие нервно-мышечного соединения, передача нервного импульса |
Крупная Белая vs ливенская |
|
|
M1GA0007600 |
MYH9 |
Внутриутробное эмбриональное развитие |
Крупная Белая vs ливенская |
|
|
7 |
MARC0045844 |
BTBD1 |
Нейрогенез |
Ливенская vs cуйтай |
|
10 |
ALGA0059118 |
BMI1 |
Внутриутробное эмбриональное развитие, гуморальный иммунный ответ, развитие мозга |
Крупная Белая vs ливенская |
|
M1GA0013933 |
CTSL |
Адаптивный иммунный ответ, иммунный ответ |
Крупная Белаяvsливенская |
|
|
M1GA0013933 |
DAPK1 |
Врожденный иммунный ответ |
Крупная Белая vs ливенская |
|
|
ASGA0047391 |
B4GALT1 |
Развитие эпителиальных клеток, острая воспалительная реакция, процесс метаболизма углеводов, биосинтеза лактозы |
Ливенская vs cуйтай |
|
|
12 |
ASGA0054177 |
MKS1 |
Развитие эмбриональной скелетной системы, общего желчного протока, мозга в эмбриональный период |
Ливенская vs cуйтай |
|
ASGA0054177 |
MPO |
Процесс метаболизма органических веществ |
Ливенская vs cуйтай |
|
|
ALGA0064395 |
SPHK1 |
Развитие кровеносных сосудов |
Ливенская vs cуйтай |
|
|
ALGA0064395 |
UBALD2 |
Связывание белков |
Ливенскаяvscуйтай |
|
|
13 |
ALGA0070164, ALGA0070180, MARC0014143, MARC0011799 |
CNTN6 |
Развитие мозга |
Крупная Белая vs ливенская |
|
MARC0021643 |
TF |
Транспорт ионов железа, клеточный гомеостаз ионов железа |
Крупная Белая vs ливенская |
|
|
ALGA0112365, ALGA0120574 |
ACKR2 |
Хемотаксис, иммунный ответ |
Крупная Белая vs ливенская |
|
|
ALGA0112365, ALGA0120574 |
CYP8B1 |
Процесс биосинтеза желчных кислот, позитивная регуляция абсорбции холестерина в кишечнике |
Крупная Белая vs ливенская |
|
|
ALGA0112365, ALGA0120574 |
KLHL40 |
Развитие волокон скелетных мышц |
Крупная Белая vs ливенская |
|
|
14 |
ALGA0078532, ALGA0078534 |
COL13A1 |
Развитие скелетной системы, врожденный иммунный ответ |
Крупная Белая vs ливенская |
|
ALGA0078532, ALGA0078534 |
NEUROG3 |
Развитие органов чувств |
Крупная Белая vs ливенская |
|
|
DRGA0014062 |
PSAP |
Регуляция процесса метаболизма липидов, рост предстательной железы |
Крупная Белая vs cутай, ЛивенскаяvsСутай |
|
|
ALGA0078532, ALGA0078534 |
TACR2 |
Регуляция сокращения гладких мышц матки, позитивная регуляция подвижности жгутиковых сперматозоидов |
Крупная Белаяvsливенская |
|
|
ALGA0081495 |
AWN |
Однократное оплодотворение |
Ливенская vs cуйтай |
|
|
ALGA0081495 |
HTRA1 |
Развитие плаценты |
Ливенская vs cуйтай |
|
|
ALGA0081495 |
PLEKHA1 |
Сперматогенез |
Ливенская vs cуйтай |
|
|
ALGA0081495 |
PSP-II |
Однократное оплодотворение |
Ливенская vs cуйтай |
|
|
15 |
ASGA0070586 |
CHRND |
Развитие ткани скелетных мышц, процессы нервной системы, развитие скелетно-мышечной мускулатуры |
Крупная Белая vs ливенская |
|
ASGA0070586 |
CHRNG |
Процесс нервной системы |
Крупная Белая vs ливенская |
|
|
ASGA0070586 |
GIGYF2 |
Постэмбриональное развитие |
Крупная Белая vs ливенская |
|
|
ALGA0087047, MARC0070665 |
HTR2B |
Морфогенез и развитие сердца, гипертрофия сердечной мышцы, сокращение гладких мышц кишечника |
Крупная Белая vs ливенская |
|
|
ASGA0070586 |
SNORC |
Развитие хряща |
Крупная Белая vs ливенская |
|
|
16 |
ALGA0091053 |
C1QTNF2 |
Врожденный иммунный ответ |
Ливенскаяvscуйтай |
|
18 |
ALGA0097092 |
CALD1 |
Ангиогенез, сокращение мышц |
Ливенская vs cуйтай |
|
ALGA0097092 |
STRA8 |
Сперматогенез |
Ливенская vs cуйтай |
Подавляющие большинство функционально идентифицированных генов (26,5%) отвечают за спектр функции в эмбриональном развитии свиней, процессами оплодотворения и формирование иммунного ответа организма (12,3%). В связи с чем дальнейшая аннотация генов была направлена на поиск их значимых взаимосвязей у других исследователей.
Так, в исследованиях X. Zhao et al. (2019), проведенных на поросятах породы ландрас, ген ACAA2 обнаружен как ключевой для процесса метаболизма липидов. Ген ACAA2 кодирует белок ацетил-КоА ацилтрансферазу 2, который катализирует последний этап бета-окисления жирных кислот в митохондриях [6–8]. В 2015 году исследования показали, что ген ACAA2 связан с энергетическим метаболизмом [9].
Ген BVES, выявленный в исследованиях M. Damon (2012) у свиней пород крупная белая и баскской, отвечает за развитие мышечных органов с высокой достоверностью (Р=1,9х10-7) [10]. Ген CDH20, расположенный в локусе хромосомы 1, ранее, в исследованиях M. Heidaritabar et al. (2023) достоверно ассоциировался с отложением жира у свиней коммерческих пород [11]. Гены GABRR1 и GABRR2 были выявлены как кандидаты, связанные со среднесуточным потреблением корма у свиней породы дюрок [12]. В данных исследованиях веб-программа DAVID ассоциировала эти гены с процессами нервной системы организма свиней, что является логичным при связывании двух анализов.
Ген TALDO1 является одним из ключевых при формировании эструса (метод управления процессом овуляции) у свиноматок крупной белой породы [13]. Ген CD247 выявлен как один из генов, нивелирующих предрасположенность к образованию пупочных грыж у свиней благодаря формированию высокого иммунологического статуса организма [14]. Ген SYPL2 обнаружен в проведенных исследованиях на крупной белой породе свиней по показателю количества живых поросят при рождении и взаимосвязан с постнатальным периодом животных [15]. Ген TEAD4 играет значимую роль при эмбриональном развитии свиньи, и его берут как основной ген при формировании и апробации новых методов, что доказывается в исследованиях многих ученых [16, 17].
В исследованиях J. Gao et al. (2016) ген MYH9 не только отвечает за эмбриональное развитие, но и является существенным фактором при заболевании свиней репродуктивно-респираторным синдромом (РРСС) [18]. Он вызывает серьезную репродуктивную недостаточность и высокий уровень поздних абортов и ранних опоросов у свиноматок, а также респираторных заболеваний и смертности молодых свиней. В настоящее время данный синдром широко признан одним из наиболее экономически значимых заболеваний в свиноводческой отрасли во всем мире.
Ген BTBD1 ранее ассоциирован с толщиной кожи товарных помесей свиней, достоверность при этом составляет 3,28х10-6 [19]. Ген BMI1 локализуется в кишечных эпителиальных клетках свиней и его используют в широких спектрах исследований, таких как созданий стволовых клеток [20] и химер [21]. Ген CTSL относится к группе катепсинов – это протеазы, которые участвуют в структурных и биохимических изменениях, происходящих во время хранения мяса после убоя [22]. Были обнаружены большие различия в активности этих ферментов среди генотипов свиней [23], при этом у крупной белой породы свиней была особенно высокая локализация данного гена в двуглавой мышце бедра [24].
Следующий ген – DAPK1, ассоциирован как один из генов-кандидатов, отвечающих за развитие скелетных мышц в организме изучаемого в настоящем исследовании вида животного [25]. В статье K. Han et al. (2022) приводят подробное описание гена B4GALT1, который является членом семейства генов бета-1,4-галактозилтрансферазы и может катализировать синтез структуры Galβ1-4GlcNAcβ1-R. Примечательно, что он показал идентичный пространственно-временной паттерн экспрессии с антигеном Lewis x, и является потенциальным модулятором на стадии прикрепления зародыша к матке свиней [26].
Ген SPHK1 является одним из ключевых ферментов в метаболизме сфинголипидов, которые предположительно влияют на формирование зародыша у свиней. И хотя роль сфинголипидов в развитии зародыша неясна, предыдущая характеристика белков в среде матки во время эстрального цикла и ранней беременности выявила четыре изоформы просапозина, которые были значительно увеличены на 13-й день беременности по сравнению с эстральным циклом и 10-м днем беременности, а это предполагает, что метаболизм сфинголипидов может играть роль в формировании зародыша и его имплантации у свиней [27]. Ген ACKR2 выявлен как потенциальный ген-кандидат, отвечающий за дерматологические заболевания и состояния, иммунологические заболевания, повреждения и аномалии организма иберийских свиней [28]. Ген KLHL40 связан с ростом и развитием мышц у свиней [29].
Период имплантации имеет решающее значение для развития концептусов на ранних стадиях беременности у свиней. В отличие от других животных, концептусы свиней претерпевают уникальную быструю трансформацию морфологии между 10-м и 12-м днями беременности, в основном из-за обширной клеточной миграции и реорганизации. Исследования показали, что быстрое удлинение зародыша приводит к увеличению его смертности, и данный вывод следует по определенному шаблону экспрессии генов [30]. В этот период синтезируемые и высвобождаемые зародышем эстрогены появляются и считаются сигналами для распознавания супоросности свиней.
Для устранения данной причины X. Zang et al. провели генетический транскриптомный анализ и выявили ген PSAP, который позволяет увеличить выживаемость зародыша на этапе имплантации у свиней [31]. Ген AWN является одним из белков-акцепторов ингибитора акрозина, ассоциированного со спермой хряков, а именно в распознавании и связывании сперматозоида с яйцеклеткой, что увеличивает процент оплодотворения [32].
Ген PLEKHA1, в исследовании Н. Wang et al. (2022), идентифицирован с показателями роста и развития четырехпородных свиней [33]. За развитие организма отвечает ген CHRND, который, в исследованиях J. Horodyska et al. (2017), ассоциирован с показателем конверсии корма у свиней породы максгро [34]. В исследованиях C. Wang et al. (2023) выявлены гены CHRND и CHRNG, которые неразрывно связаны с развитием и структурой мышц [35]. Ген STRA8 необходим для таких процессов, как хромосомная когезия, образование синаптонемных комплексов и рекомбинация, и действует как ген-контролер мейоза. В исследованиях X. Wang et al. (2014), на хряках породы мейшань, проведено полное клонирование данного гена для его более подробной характеристики [36].
Заключение
Проведенные исследования по выявлению генетической дифференциации статистическим методом FST показали высокое однообразие между крупной белой породой и китайской породой сутай (FST =0,187), наименьшее однообразие – между крупной белой породой свиней и локальной российской ливенской породой (FST =0,070).
Проведенная аннотация выявленных 222 генов показала функциональную значимость 49 генов по следующему распределению: 6 генов по группе пород свиней крупной белой породы и ливенской, 26 генов – крупной белой породы и сутай и 18 – ливенской породы и китайской породой свиней сутай. Подавляющие большинство функционально идентифицированных генов отвечают за спектр функции в эмбриональном развитии свиней, включая процессы оплодотворения – 26,5%, и за формирование иммунного ответа организма – 12,3%. В связи с чем дальнейшая аннотация генов была направлена на поиск их значимых взаимосвязей у других исследователей, представленных в тексте настоящей статьи.
Результаты данных исследований можно использовать при изучении сходимости пород, выявления их исторического становления, написания научных статей, учебных пособий и практиковать на курсах повышения квалификации.
Исследование выполнено при финансовой поддержке Российского научного фонда, проект №23-46-00014
Литература
- Указ Президента Российской Федерации от 28 февраля 2024 года №145 «О Стратегии научно-технологического развития Российской Федерации».
- Wright S. The interpretation of population structure by F-statistics with special regard to systems of mating. Evolution, 1965. Vol. 19. P. 395–420. DOI: 10.1111/j.1558-5646.1965.tb01731.x.
- Gizaw S. Population structure, genetic variation and morphological diversity in indigenous sheep of Ethiopia/S. Gizaw, J. van Arendonk, H. Komen, J.J. Windig, O. Hanotte. Anim. Genet., 2007. Vol. 38. P. 621–628. DOI: 10.1111/j.1365-2052.2007.01659.x.
- Colino-Rabanal V.J. Human and ecological determinants of the spatial structure of local breed diversity/V.J. Colino-Rabanal, R. Rodríguez-Díaz, M.J. Blanco-Villegas, S.J. Peris, M. Lizana. Sci. Rep., 2018. Vol. 8. P. 6452. DOI: 10.1038/s41598-018-24641-3.
- Berthouly C. How does farmer connectivity influence livestock genetic structure? a case-study in a vietnamese goat population/C. Berthouly, D.D. Ngoc, S. Thévenon, D. Bouchel, C. Van, T.N. Danes, V. Grosbois, H.H. Thanh, C.V. Chi, J.-C. Maillard. Mol. Ecol., 2009. Vol. 18. P. 3980–3991. DOI: 10.1111/j.1365-294X.2009.04342.x.
- Cortellari M. The climatic and genetic heritage of Italian goat breeds with genomic SNP data/M. Cortellari, M. Barbato, A. Talenti, A. Bionda, A. Carta, R. Ciampolini, E. Ciani, A. Crisa, S. Frattini, E. Lasagna. Sci. Rep., 2021. Vol. 11. P. 10986. DOI: 10.1038/s41598-021-89900-2.
- Wang Q.Q. Cloning of goose acetyl-coenzyme a acyltransferase 2 and its expression pattern in the development of fatty liver/Q.Q. Wang, B. Yang, L.L. Xia, X.X. Sun, T.Y. Geng, D.Q. Gong. J. Anim. Vet. Sci., 2016. Vol. 47. P. 700–708. DOI: 10.11843/j.issn.0366-6964.2016.04.009.
- Sodhi S.S. An approach to identify SNPs in the gene encoding acetyl-CoA acetyltransferase-2 (ACAT-2) and their proposed role in metabolic processes in pig/S.S. Sodhi, M. Ghosh, K.D. Song, N. Sharma, J.H. Kim, N.E. Kim, S.J. Lee, C.W. Kang, S.J. Oh, D.K. Jeong. PLoS ONE, 2014. Vol. 9. P. e102432. DOI: 10.1371/journal.pone.0102432.
- Miltiadou D. Variants in the 3’ untranslated region of the ovine acetyl-coenzyme A acyltransferase 2 gene are associated with dairy traits and exhibit differential allelic expression/D. Miltiadou, A.C. Hager-Theodorides, S. Symeou, C. Constantinou, A. Psifidi, G. Banos, O. Tzamaloukas. J. Dairy Sci., 2017. Vol. 100. P. 6285–6297.
- Wang C. Influence of delipation on the energy metabolism in pig parthenogenetically activated embryos/C. Wang, Y. Niu, D. Chi, Y. Zeng, H. Liu, Y. Dai, J. Li. Reprod. Domest. Anim., 2015. Vol. 50. P. 826–833. DOI: 10.1111/rda.12596.
- Damon M. Comparison of muscle transcriptome between pigs with divergent meat quality phenotypes identifies genes related to muscle metabolism and structure/M. Damon, J. Wyszynska-Koko, A. Vincent, F. Hérault, B. Lebret. PLoS One, 2012. Vol. 7(3). P. e33763. DOI: 10.1371/journal.pone.0033763.
- Heidaritabar M. Genome-wide association studies of additive and dominant effects for body composition traits in commercial crossbred Pietrain pigs/M. Heidaritabar, M.C.A.M. Bink, E. Dervishi, P. Charagu, A. Huisman, G.S. Plastow. J. Anim. Breed Genet., 2023. Vol. 140 P. 413–430. DOI: 10.1111/jbg.12768.
- Do D.N. Genome-wide association study reveals genetic architecture of eating behavior in pigs and its implications for humans obesity by comparative mapping/D.N. Do, A.B. Strathe, T. Ostersen, J. Jensen, T. Mark, H.N. Kadarmideen. PLoS One, 2013. Vol. 8(8). P. e71509. DOI: 10.1371/journal.pone.0071509.
- Li C. Identification of estrus in sows based on salivary proteomics/C. Li, C. Song, K. Qi, Y. Liu, Y. Dou, X. Li, R. Qiao, K. Wang, X. Han, X. Li. Animals (Basel), 2022. Vol. 12(13). P. 1656. DOI: 10.3390/ani12131656.
- Souza M.R. Transcriptome analysis identifies genes involved with the development of umbilical hernias in pigs/M.R. Souza, A.M.G. Ibelli, I.R. Savoldi, M.E. Cantão, J.O. Peixoto, M.A.Z. Mores, J.S. Lopes, L.L. Coutinho, M.C. Ledur Souza. PLoS One, 2020. Vol. 15(5). P. e0232542. DOI: 10.1371/journal.pone.0232542.
- Freitas P.H.F. Genomic predictions and GWAS for heat tolerance in pigs based on reaction norm models with performance records and data from public weather stations considering alternative temperature thresholds/P.H.F. Freitas, J.S. Johnson, F. Tiezzi, Y. Huang, A.P. Schinckel, L.F. Brito. J. Anim. Breed Genet., 2024. Vol. 141(3). P. 257–277. DOI: 10.1111/jbg.12838.
- Cao S. Specific gene-regulation networks during the pre-implantation development of the pig embryo as revealed by deep sequencing/J. Han, J. Wu, Q. Li, S. Liu, W. Zhang, Y. Pei, X. Ruan, Z. Liu, X. Wang, B. Lim, N. Li. BMC Genomics, 2014. Vol. 15(1). P. 4. DOI: 10.1186/1471-2164-15-4.
- Natsuko E. The necessity of TEAD4 for early development and gene expression involved in differentiation in porcine embryos/E. Natsuko, T. Kazuki, S. Yuriko, S. Ken. Journal of Reproduction and Development, 2019. Vol. 65(4). P. 361–368. DOI: 10.1262/jrd.2018-120.
- Gao J. MYH9 is an essential factor for porcine reproductive and respiratory syndrome virus infection/J. Gao, S. Xiao, Y. Xiao. Sci. Rep., 2016. Vol. 6. P. 25120. DOI: 10.1038/srep25120.
- Wang H. Genome-wide identification of quantitative trait loci and candidate genes for seven carcass traits in a four-way intercross porcine population/H. Wang, X. Wang, Y. Yang, Y. Zhu, S. Wang, Q. Chen, D. Yan, X. Dong, M. Li, S. Lu. BMC Genomics, 2024. Vol. 25(1). P. 582. DOI: 10.1186/s12864-024-10484-y.
- Li X.G. LGR5 and BMI1 increase pig intestinal epithelial cell proliferation by stimulating WNT/β-catenin signaling/X.G. Li, Z. Wang, R.Q. Chen, H.L. Fu, C.Q. Gao, H.C. Yan, G.X. Xing, X.Q. Wang. Int. J. Mol. Sci., 2018. Vol. 19(4). P. 1036. DOI: 10.3390/ijms19041036.
- Xuan Y. Human and pig pluripotent stem cells: From cellular products to organogenesis and beyond/Y. Xuan, B. Petersen, P. Liu. Cells, 2023. Vol. 12. P. 2075. DOI: 10.3390/cells12162075.
- Flores M. Effect of brine thawing/salting on endogenous enzyme activity and sensory quality of Iberian dry-cured ham/M. Flores, M.C. Aristoy, T. Antequera, J.M. Barat, F. Toldrá. Food Microbiol., 2012. Vol. 29(2). P. 247–254. DOI: 10.1016/j.fm.2011.06.011.
- Hernandez P. Antioxidant, lipolytic and proteolytic enzyme activities in pork meat from different genotypes/P. Hernandez, L. Zomeno, B. Arino, A. Blasco. Meat Sci., 2004. Vol. 66(3). P. 525–529. DOI: 10.1016/S0309-1740(03)00155-4.
- Balatsky V. Polymorphisms of the porcine cathepsins, growth hormone-releasing hormone and leptin receptor genes and their association with meat quality traits in Ukrainian Large White breed/V. Balatsky, I. Bankovska, R.N. Pena, A. Saienko, T. Buslyk, S. Korinnyi, O. Doran. Mol. Biol. Rep., 2016. Vol. 43(6). P. 26–517. DOI: 10.1007/s11033-016-3977-z.
- Feng L. the landscape of accessible chromatin and developmental transcriptome maps reveal a genetic mechanism of skeletal muscle development in pigs/L. Feng, J. Si, J. Yue, M. Zhao, W. Qi, S. Zhu, J. Mo, L. Wang, G. Lan, J. Liang. Int. J. Mol. Sci., 2023. Vol. 24(7). P. 6413. DOI: 10.3390/ijms24076413.
- Han K. Lewis x-carrying O-glycans are candidate modulators for conceptus attachment in pigs/K. Han, Y. Yue, W. Wang, F. Wang, W. Chai, S. Zhao, M. Yu. Biol. Reprod., 2023. Vol. 108(2). P. 292–303. DOI: 10.1093/biolre/ioac204.
- Miles J.R. Mechanisms regulating the initiation of porcine conceptus elongation/J.R. Miles, S.C. Walsh, L.A. Rempel, A.K. Pannier. Mol. Reprod. Dev., 2023. Vol. 90(7). P. 646–657. DOI: 10.1002/mrd.23623.
- Fernández-Barroso M.Á. Understanding the role of myoglobin content in Iberian pigs fattened in an extensive system through analysis of the transcriptome profile/M.A. Fernández-Barroso, J.M. García-Casco, Y. Núñez, L. Ramírez-Hidalgo, G. Matos, M. Muñoz. Anim. Genet., 2022. Vol. 53(3). P. 352–367. DOI: 10.1111/age.13195.
- Liufu S. Transcriptome analysis reveals the age-related developmental dynamics pattern of the longissimus dorsi muscle in Ningxiang pigs/S. Liufu, Q. Lan, X. Liu, B. Chen, X. Xu, N. Ai, X. Li, Z. Yu, H. Ma. Genes (Basel), 2023. Vol. 14(5). P. 1050. DOI: 10.3390/genes14051050.
- Niemann H. Alterations of expression of developmentally important genes in preimplantation bovine embryos by in vitro culture conditions: Implications for subsequent development/H. Niemann, C. Wrenzycki. Theriogenology, 2000. Vol. 53(1). P. 21–34. DOI: 10.1016/s0093-691x(99)00237-x.
- Zang X. Global transcriptomic analyses reveal genes involved in conceptus development during the implantation stages in pigs/X. Zang, T. Gu, Q. Hu, Z. Xu, Y. Xie, C. Zhou, E. Zheng, S. Huang, Z. Xu, F. Meng, G. Cai, Z. Wu, L. Hong. Front Genet., 2021. Vol. 12. P. 584995. DOI: 10.3389/fgene.2021.584995.
- Sanz L. Boar spermadhesins AQN-1 and AWN are sperm-associated acrosin inhibitor acceptor proteins/L. Sanz, J.J. Calvete, V. Jonáková, E. Töpfer-Petersen. FEBS Lett, 1992. Vol. 300(1). P. 63–66. DOI: 10.1016/0014-5793(92)80164-c.
- Wang H. Genome-wide association study of growth traits in a four-way crossbred pig population/H. Wang, X. Wang, M. Li, H. Sun, Q. Chen, D. Yan, X. Dong, Y. Pan, S. Lu. Genes, 2022. Vol. 13. P. 1990. DOI: 10.3390/genes13111990.
- Horodyska J. Genome-wide association analysis and functional annotation of positional candidate genes for feed conversion efficiency and growth rate in pigs/J. Horodyska, R.M. Hamill, P.F. Varley, H. Reyer, K. Wimmers. PLoS One, 2017. Vol. 12(6). P. e0173482. DOI: 10.1371/journal.pone.0173482.
- Wang C. An analysis of a transposable element expression atlas during 27 developmental stages in porcine skeletal muscle: Unveiling molecular insights into pork production traits/C. Wang, B. Lei, Y. Liu. Animals (Basel), 2023. Vol. 13(22). P. 3581. DOI: 10.3390/ani13223581.
- Wang X. Cloning and expression characteristics of the pig Stra8/X. Wang, T. Chen, C. Song, B. Gao, Y. Zhang. Gene. Int. J. Mol. Sci., 2014. Vol. 15. P. 12480–12494. DOI: 10.3390/ijms150712480.








