В 2025 году журналу
ИСПОЛНИТСЯ
95 ЛЕТ!
ЧИТАЙТЕ
Материалы к юбилею >>>

РУБРИКИ
 Племенное дело |
 Крупный план |
 Актуально |
 Корма |
 Техническая политика |
 Событие |
 Ветеринария |
 Эксклюзивное интервью |
 Статистика |
 Выставки |
Творчество
наших читателей
ЧИТАЙТЕ
Автор С.И. КАЛИНИЧЕНКО
ГИМН СВИНЬЕ >>>
СТАТЬИ ИЗ НОМЕРА
DOI: 10.37925/0039-713X-2024-6-39-44
УДК 636.4.033
ГЕНОМНАЯ ОЦЕНКА ПРИЗНАКА КОНВЕРСИИ КОРМА СВИНЕЙ ПОРОДЫ ДЮРОК
А.А. БЕЛОУС, кандидат биолог. наук, старший научный сотрудник, e-mail: belousa663@gmail.com, П.И. ОТРАДНОВ, младший научный сотрудник, Н.А. ЗИНОВЬЕВА, доктор биолог. наук, профессор, академик РАН, Федеральный исследовательский центр животноводства – ВИЖ имени академика Л.К. Эрнста
В настоящее время внедрение геномной оценки всех видов сельскохозяйственных животных и объектов товарной аквакультуры является актуальной и необходимой целью научной работы.
В данной статье представлены результаты полногеномных ассоциативных исследований по конверсии корма на основании двух основных методологий BLUP – BLUP Animal Model и genomic BLUP. Приведен пример каталогизации параметров геномной оценки по эффективности использования корма и характеристикам кормового поведения свиней породы дюрок для внедрения в практику. Выявлено пять значимых генов, по которым в дальнейшем будут разработаны тест-системы.
Ключевые слова: геномная оценка, оценка племенной ценности, полногеномное ассоциативное исследование, конверсия корма, свиньи породы дюрок.
Genomic evaluation of feed conversion trait in Duroc pigs
А.А. BELOUS, candidate of biological sciences, senior researcher, e-mail: belousa663@gmail.com, P.I. OTRADNOV, junior researcher, N.A. ZINOVIEVA, doctor of biological sciences, professor, academician of the RAS, Federal Research Center for Animal Husbandry named after academy member L.K. Ernst
Currently, the implementation of genomic evaluation in all types of farm animals and commercial aquaculture objects is an urgent and necessary goal of scientific work.
This paper presents the results of genome-wide association studies on feed conversion based on two main BLUP methodologies – BLUP Animal Model and genomic BLUP. An example of genomic evaluation parameters cataloging by feed efficiency and feeding behavior characteristics in Duroc pigs for implementation and application in practical direction is given. Five significant genes were identified, for which test systems will be developed in the future.
Key words: genomic evaluation, breeding value assessment, full genome association study, feed conversion, Duroc pigs.
Введение
На корма приходится около 65% общих затрат в современном свиноводстве, и характеристики эффективности использования корма (Feed efficiency – FE) имеют решающее значение [1]. Оптимизация этого показателя приводит к снижению себестоимости, тем самым способствуя снижению конечной стоимости продукции [2]. В связи с этим программы селекции для улучшения FE осуществляются уже много лет и данный показатель оценивается по четырем признакам – коэффициенту конверсии корма, среднесуточному привесу, среднесуточному потреблению корма и прогнозируемому остаточному потреблению корма [3, 4]. Для оптимизации вышеперечисленных признаков важно понять молекулярный механизм и генетический базис, лежащий в основе характеристик, связанных с FE.
За последние десятилетия были обнаружены сотни локусов количественных признаков (QTL), связанных со сложными признаками у свиней, включая признаки, характеризующие FE. Среди них 346, 618, 96 и 96 QTL для FCR, ADG, ADFI и RFI соответственно были идентифицированы в популяциях свиней разных пород. Однако механизм их молекулярной регуляции остается в значительной степени неизвестным, в связи с чем разработка программ геномной оценки свиней по хозяйственно полезным признакам является затруднительной, так как сочетает в себе комплекс мероприятий «от фенотипа – к генотипу».
В России впервые геномная оценка была создана и апробирована на КРС молочного направления продуктивности [5, 6]. В свиноводстве на хряках породы дюрок разработка геномной селекции по показателям конверсии корма и кормового поведения началась в 2017 году [7]. Она была изучена с разных сторон – от расчета параметров на основании первичных данных, полученных с автоматических кормовых станций [8], до полногеномных ассоциативных исследований (GWAS) [9, 10] и апробации селекционного индекса [11].
Изучив статистические и молекулярные аспекты упомянутых характеристик данной породы, мы пришли к выводу, что для повышения достоверности результатов необходимо руководствоваться подходами, учитывающими обе эти компоненты. В связи с чем сочетание оценки племенной ценности методами BLUP (BLUP AM и GBLUP) и обработки геномной информации в рамках методологии GWAS видится перспективным подходом для разработки программы геномной оценки свиней породы дюрок по значимым хозяйственно полезным признакам.
Цель исследования – разработка концепции геномной селекции свиней породы дюрок по признакам конверсии корма на основании двух подходов в рамках методологии BLUP и полногеномного анализа ассоциаций.
Материал и методы исследований
Исследования проводились на свиньях коммерческой породы дюрок (n=800), проходящих откорм на автоматических кормовых станциях MLP-RAP (Schauer Agrotronic AG, Швейцария) в ООО СГЦ «Топ Ген» в 2017–2020 годах.
В статье представлены следующие сокращения и условные обозначения:
▪ BLUP AM (BLUP Animal Model) – оценка племенной ценности с учетом родословной;
▪ GBLUP (genomic BLUP) – оценка племенной ценности с учетом данных генотипирования;
▪ FCR (кг/кг) – конверсия корма;
▪ ADG (г) – среднесуточный прирост;
▪ ADFI (г/сут.) – среднесуточное потребление корма;
▪ BW0 (кг) – начальная живая масса;
▪ BW1 (кг) – конечная живая масса;
▪ FR (г/мин.) – скорость потребления корма;
▪ FPV (г/ед.) – среднее количество потребленного корма за посещение кормовой станции;
▪ NVD (ед.) – количество посещений кормовой станции;
▪ TPD (мин.) – среднее время, проведенное на кормовой станции в сутки;
▪ TPV (мин/ед.) – среднее время посещения кормовой станции.
Описательная характеристика признаков в исследованной выборке представлена в таблице 1.
Таблица 1. Описательные статистические параметры исследованных признаков
|
Признак |
Параметр |
|||
|
М±m |
Минимум…максимум |
Коэффициент вариации, % |
Картина распределения |
|
|
FCR, кг/кг |
2,45±0,01 |
1,24…4,54 |
16,27 |
|
|
ADG, г |
993,41±3,37 |
579,17…1507,69 |
12,79 |
|
|
ADFI, г/сут. |
2408,36±9,23 |
1140,92…3951,19 |
14,43 |
|
|
BW0, кг |
37,09±0,17 |
18…65 |
17,46 |
|
|
BW1, кг |
110,1±0,29 |
79,9…157 |
9,77 |
|
|
FR, г/мин. |
35,58±0,2 |
16,56…74,49 |
21,45 |
|
|
FPV, г |
310,92±3,58 |
82,28…798,94 |
43,30 |
|
|
NVD, ед. |
9,1±0,08 |
3,5…27,35 |
31,19 |
|
|
TPD, мин. |
71,07±0,34 |
39,81…139,91 |
18,09 |
|
|
TPV, мин. |
9,34±0,11 |
2,25…25,98 |
44,28 |
|
Исследованные признаки демонстрировали умеренный уровень вариации (CV=9,77…44,28%). Наибольшей вариативностью характеризовались признаки NVD, FPV и TPV (31,19%, 43,30%, 44,28% соответственно), наименьшей – BW1, ADG, ADFI (9,77%, 12,79%, 14,43% соответственно). Картина распределения для признаков ADG, ADFI, BW0, BW1, FR, NVD и TPD соответствовала нормальному распределению. Признаки FCR, FPV, TPV демонстрировали логнормальное распределение (смещенность вершины относительно среднего).
Оценка племенной ценности животных по всем признакам не потребовала дополнительных преобразований и проводилась с применением методологий BLUP Animal Model, GBLUP [11].
Для оценки влияния случайных средовых факторов на исследуемые признаки был сформирован рандомизированный эффект DFSM («Дата постановки на откорм – номер кормовой станции – возраст в неделях»). Подобная классификация в рамках используемой методологии применяется для определения изменчивости, вызванной случайными эффектами среды и исключения ее воздействия на итоговую оценку племенной ценности животного. Оценка влияния факторов в линейной модели производилась с помощью библиотеки lme4 версии 1.1-32 в программной среде R версии 4.2.3. Модель формировалась по двум критериям – объяснимость воздействия факторов на признак и достоверность влияния факторов α≥0,95.
Таким образом, смешанные математические модели оценки признаков имели вид:
ADFI = μ + DFSM + b1FR + b2FPV + b3BWo + a + e;
FR = μ + DFSM + b1TPV + b2FPV + b3BWo + a + e;
TPV = μ + DFSM + b1TPV + a + e;
NVD
FPV = μ + DFSM + b1TPV + b2FPV + a + e;
TPD
FCR = μ + DFSM + b1ADFI + a + e;
ADG = μ + DFSM + b1ADFI + a + e;
BW0 = μ + YMB + b1Age0 + a + e;
BW1 = μ + DFSM + b1ADFI + b2ADG + a + e;
где μ – популяционное среднее, DFSM – рандомизированный эффект среды, BW0 – фиксированный эффект живой массы при постановке на откорм, a – рандомизированный генетический эффект животного (собственно, оценка племенной ценности), e – эффект неучтенных в модели факторов, b1-b3 – фиксированные коэффициенты регрессии оцениваемого признака на соответствующие эффекты.
В общем матричном виде уравнение смешанной модели BLUP AM решается, исходя из уравнения:
где X, Z – матрицы распределения наблюдений (записей о фенотипах в группах эффектов), X’, Z’ – транспонированные матрицы X и Z, A-1 – обратная матрица родства, – степень влияния на признак негенетических эффектов,
– коэффициент наследуемости.
В рамках методологии GBLUP обратная матрица родства A-1 заменяется обратной матрицей геномного сходства G-1:
где M – матрица содержания SNP-файла (закодированная зиготность SNP, где 0 – гомозиготность AA, 1 – гетерозиготность AB, 2 – гомозиготность BB, 5 – данные об SNP отсутствуют), M’ – транспонированная матрица M. pi=(1-α), где α – достоверность ассоциации i-го SNP с фенотипическими значениями оцениваемого признака.
В качестве материала для молекулярно-генетических исследований использовали пробы ткани (ушные выщипы). Выделение ДНК проводили с помощью наборов для выделения геномной ДНК серии «ДНК-Экстран» (ЗАО «Синтол», Россия) в соответствии с протоколом фирмы-производителя. Концентрацию двухцепочечной ДНК определяли посредством флуориметра Qubit 2.0 (Invitrogen/Life Technologies, США). Для определения качества ДНК измеряли соотношение OD260/OD280 (cпектрофотометр NanoDrop 8000, Thermo Fisher Scientific, США). Для анализа использовали ДНК с OD260/OD280=1,6–1,8. Кроме того, качество ДНК оценивали посредством гель-электрофореза в 1%-ном агарозном геле. Полногеномное генотипирование производили с применением высокоплотных ДНК-чипов PorcineHD Genotyping BeadChip (платформа GeneSeek Genomic Profiler, Neogene, США), содержащим 66 763 SNP.
Анализ полногеномных ассоциаций осуществлялся с помощью программы PLINK 1.90 с учетом популяционной стратификации. Контроль качества и фильтрацию данных генотипирования для каждого SNP и каждого образца выполняли, применяя фильтры call rate по всем исследуемым SNP для индивидуального образца не ниже 90% (-mind 0.10), call rate для каждого из исследованных SNP по всем генотипированным образцам – не ниже 90% (-geno 0.10). Частота встречаемости минорных аллелей (MAF) – ≥0,05. Отклонение генотипов по SNP от распределения по Харди-Вайнбергу в совокупности протестированных образцов – ≤0,001 (-hwe 1e-3).
Для поиска генов-кандидатов, локализованных в области идентифицированных SNP, использовали геномный ресурс Sscrofa11.1 (https://www.ncbi.nlm.nih.gov/datasets/genome/GCF_000003025.6/).
Функциональные аннотации и выявление анализа обогащения генов выполняли с привлечением базы данных DAVID (https://david.ncifcrf.gov/summary.jsp) и базы данных Animal QTL (https://www.animalgenome.org/cgi-bin/QTLdb/index).
Результаты
Оценки племенной ценности по двум рассматриваемым методологиям рассчитывались на 800 головах свиней, имеющих в наличии родословную и генотипы с чипов высокой плотности.
С целью выявления взаимосвязанности признаков был проведен анализ коэффициентов генетической и фенотипической корреляций. На генетическом уровне была обнаружена очень тесная степень взаимосвязи (как отрицательной, так и положительной) между парами признаков ADG-FCR (-0,98), ADFI-TPD (0,92), NVD-ADFI (0,76) и NVD-TPD (0,77). На фенотипическом же уровне наибольшая теснота взаимосвязи характеризовала пары признаков NVD-FPV (-0,89), TPV-NVD (-0,85), TPV-FPV (0,80).
Высокая степень взаимосвязанности признаков на генетическом уровне, как правило, свидетельствует об общности генетических факторов, таких как происхождение, влияющих на их изменчивость. Так, перспектива селекции ADG и FCR демонстрирует желательную тенденцию: увеличение генетической количественной компоненты (собственно, племенной ценности) признака ADG приведет к снижению таковой для FCR, то есть при больших значениях среднесуточного прироста произойдет сокращение потребления корма на 1 кг прироста. Также справедливо и обратное.
Рис. 1. Пример каталогизации параметров геномной оценки по эффективности использования корма и кормового поведения свиней породы дюрок
Для практического применения полученных результатов разработан макет визуализации для последующего создания каталога лучших животных по фактическим данным и оценкам племенной ценности. Так, на рисунке 1 продемонстрирован хряк породы дюрок, имеющий наивысшие результаты оценки по конверсии корма (BLUP AM – -0,16; GBLUP – -0,30). При этом он также является улучшателем по признакам среднесуточного прироста и среднесуточного потребления корма. Незначительная разница наблюдается в оценках по характеристикам количества посещений фидлота (кормовой станции), потребления корма (ADFI, FPV), начальной и конечной живой массы.

Рис. 2. Полногеномный анализ ассоциаций с признаком FCR на основании (сверху вниз): 1 – оценок BLUP Animal Model, 2 – оценок genomic BLUP
Анализ полногеномных ассоциаций с признаком FCR показал наличие SNP, имеющих совпадение по обеим методологиям (рис. 2). Всего было выявлено 31 SNP на 9 из 18 хромосомах.
Таблица 2. Структурная аннотация выявленных общих SNP по рассматриваемым методологиям признака конверсии корма
|
№ хромосомы |
Имя SNP |
Позиция |
P-value |
Модель оценки |
ГенПротяженность |
|
5 |
ALGA0032795 |
71 424 521 |
2.57E-06 |
BLUP AM |
SLC2A1371294999…71673853 REDIC171185567…71265935 |
|
6.20E-06 |
GBLUP |
||||
|
ASGA0026250 |
71 448 545 |
2.57E-06 |
BLUP AM |
||
|
6.20E-06 |
GBLUP |
||||
|
WU_10.2_5_92728950 |
88 382 736 |
9.01E-15 |
GBLUP |
FGD688087032…88229807 NR2C188231180…88278803 NDUFA1288292760...88317111 TMCC388409560…88682328 |
|
|
2.16E-09 |
BLUP AM |
||||
|
WU_10.2_5_108425230 |
103 167 883 |
7.92E-09 |
GBLUP |
NAV3103013798…103863222 |
|
|
3.59E-06 |
BLUP AM |
||||
|
7 |
ASGA0031167 |
10 584 379 |
1.56E-06 |
BLUP AM |
CD8310346122…10385968 |
|
6.32E-06 |
GBLUP |
||||
|
ALGA0039273 |
20 105 440 |
5.06E-08 |
GBLUP |
CARMIL120036430...20353431 CMAH19879367…19917761 |
|
|
5.92E-08 |
BLUP AM |
||||
|
8 |
ALGA0049543 |
124 919 219 |
8.79E-09 |
BLUP AM |
BMPR1B124536648...125035841 PDLIM5125044027…125259969 |
|
4.44E-08 |
GBLUP |
||||
|
WU_10.2_8_138718577 |
129 332 896 |
1.94E-06 |
BLUP AM |
SNCA129247724…129388307 MMRN1129113951...129173413 |
|
|
8.83E-06 |
GBLUP |
||||
|
11 |
WU_10.B_11_6755744 |
7 000 384 |
1.49E-06 |
BLUP AM |
KATNAL16998003...7064986 HMGB17195556...7321071 TEX267571787...7593557 MEDAG7545400...7562741 |
|
3.77E-06 |
GBLUP |
||||
|
ALGA0060596 |
7 569 688 |
1.01E-06 |
BLUP AM |
USPL17321187...7361280 ALOX5AP7371328...7438111 HSPH17733874...7759528 |
|
|
4.45E-06 |
GBLUP |
||||
|
WU_10.2_11_8706929 |
9 004 017 |
3.90E-07 |
BLUP AM |
N4BP2L18860777...8889982 BRCA28805950...8858128 PDS5B9014694...9223035 ZAR1L8795478...8805876 N4BP2L28891053...8997810 |
|
|
3.03E-06 |
GBLUP |
||||
|
WU_10.2_11_8900679 |
9 145 811 |
3.90E-07 |
BLUP AM |
||
|
3.03E-06 |
GBLUP |
||||
|
ASGA0050172 |
20 426 453 |
2.29E-08 |
GBLUP |
HTR2A20555939...20620225 |
|
|
4.91E-07 |
BLUP AM |
||||
|
ALGA0062301 |
49 061 964 |
2.65E-06 |
GBLUP |
KCTD1249025134...49033208 ACOD149077152…49088218 CLN549105822...49128425 FBXL349131663...49151380 MYCBP249163969…49432865 |
|
|
3.84E-06 |
BLUP AM |
||||
|
15 |
DRGA0015042 |
27 445 077 |
1.75E-06 |
BLUP AM |
CNTNAP526875305...27740340 |
|
2.23E-06 |
GBLUP |
||||
|
WU_10.2_15_142651053 |
128 847 114 |
1.76E-06 |
GBLUP |
COL4A3128611640...128763331 MFF128768441...128799355 TM4SF20128802438...128814201 AGFG1128898806...128980782 |
|
|
3.46E-06 |
BLUP AM |
||||
|
17 |
M1GA0021944 |
34 100 230 |
9.38E-10 |
GBLUP |
TMEM74B34099511...34107077 FKBP1A33910047…33935212 SNPH33986511…34028610 SDCBP233967075…33985975 RAD21L134035526…34064215 PSMF134118324...34164723 RSPO434268702...34310073 |
|
3.45E-07 |
BLUP AM |
||||
|
H3GA0048762 |
35 179 741 |
5.12E-07 |
GBLUP |
DEFB12535003914...35010677 DEFB11535029031...35034148 DEFB11635063977...35067003 DEFB12135154177...35155772 DEFB12335185257...35190686 DEFB12435200560...35205507 REM135208344...35218771 HM1335248575...35288431 ID135316518...35317742 |
|
|
3.20E-06 |
BLUP AM |
||||
|
WU_10.2_17_46055657 |
40 639 247 |
1.54E-07 |
BLUP AM |
MANBAL40420745...40449657 SRC40470970...40524862 NNAT40625990…40629523 BLCAP40603977...40633613 CTNNBL140809932...40948844 |
|
|
3.93E-07 |
GBLUP |
||||
|
18 |
ASGA0090634 |
7 598 438 |
7.22E-07 |
GBLUP |
EPHB67346793…7419859 PRSS27443327...7446625 |
|
4.16E-06 |
BLUP AM |
||||
|
ALGA0121880 |
7 775 625 |
5.56E-06 |
GBLUP |
MGAM27767831…7863717 |
|
|
9.23E-06 |
BLUP AM |
Примечание: полужирным шрифтом выделен ген, внутри которого локализован выявленный SNP-маркер
В таблице 2 представлен список SNP, структурная аннотация которых показала их наличие или внутри генов, или сцепления с данным участком хромосомы. Выявлен 61 ген-кандидат на 7 хромосомах, из них 10 – внутри которых локализованы SNP. Количество генов с высоким значением достоверности ассоциации, идентифицированных по общему списку для двух методологий BLUP, уточняет и улучшает полученные результаты по оценкам племенной ценности свиней породы дюрок.
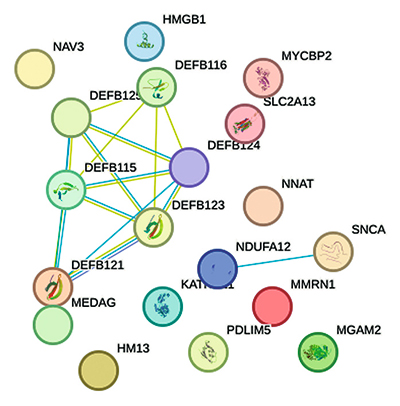
Рис. 3. Функциональная кластеризация выявленных генов-кандидатов
Функциональная аннотация показала наличие 27 генов, кластеризуемых на 19 узлах и имеющих следующее обогащение по биологическому процессу (рис. 3):
▪ защитный ответ на бактериальную микрофлору (GO:0042742) и врожденный иммунный ответ организма (GO:0045087) – шесть генов (DEFB125, DEFB116, DEFB123, DEFB115, DEFB121, DEFB124) и семь генов (DEFB125, DEFB116, DEFB123, DEFB115, DEFB121, DEFB124, HMGB1) соответственно.
▪ реакция организма на стресс (GO:0006950) – 10 генов (DEFB125, DEFB116, DEFB123, DEFB115, DEFB121, DEFB124, HMGB1, HM13, MMRN1, NDUFA12).
Для понимания достоверности влияния идентифицированных генов, следующий этап аннотации провели с использованием базы данных QTL свиней (https://www.animalgenome.org/cgi-bin/QTLdb/SS/search<), таблица 3.
Таблица 3. Функциональная аннотация генов-кандидатов по признаку конверсии корма свиней породы дюрок
|
Ген |
DAVID |
Pig data base |
|
SLC2A13 |
Позитивная регуляция образования бета-амилоида |
Содержание внутримышечного жира (QTL:224714; QTL:224704; QTL:224715; QTL:224706; QTL:224718) |
|
NDUFA12 |
Реакция на окислительный стресс |
– |
|
NAV3 |
Развитие нервной системы, нейрогенез |
– |
|
CARMIL1 |
– |
Обхват груди (QTL:286690) |
|
BMPR1B |
– |
Строение задних ног (QTL:125675), вес помета (QTL:213847; QTL:213852) и масса желудка (QTL:264171) |
|
PDLIM5 |
Развитие сердца и мышечной структуры |
Содержание внутримышечного жира (QTL:21961) |
|
SNCA |
Регуляция гибели нейронов |
– |
|
MMRN1 |
Свертывание крови |
– |
|
KATNAL1 |
Сперматогенез |
Время, проведенное в фидлоте (QTL:22411; QTL:22412) |
|
HMGB1 |
Адаптивный и врожденный иммунный ответ |
– |
|
TEX26 |
Время, проведенное в фидлоте (QTL:22382) |
|
|
MEDAG |
Позитивная регуляция дифференцировки жировых клеток |
– |
|
BRCA2 |
Пупочная грыжа (QTL:284951) |
|
|
KCTD12 |
Пигментация радужной оболочки глаз (QTL:213117) |
|
|
MYCBP2 |
Регуляция метаболических процессов |
Пигментация радужной оболочки глаз (QTL:213059; QTL:213093; QTL:213109) |
|
COL4A3 |
Средняя толщина шпика (QTL:28084) |
|
|
DEFB125 |
Защитная реакция на бактериальную инфекцию, врожденный иммунный ответ |
– |
|
DEFB116 |
||
|
DEFB123 |
||
|
DEFB115 |
||
|
DEFB121 |
||
|
DEFB124 |
||
|
HM13 |
Внутриутробное эмбриональное развитие |
– |
|
NNAT |
Развитие мозга, положительный ответ на секрецию инсулина |
– |
|
BLCAP |
– |
Время, проведенное в фидлоте (QTL:22422) |
|
CTNNBL1 |
– |
Шпик и огузок (QTL:18537), толщина подкожной жировой клетчатки (QTL:18536) |
|
MGAM2 |
Процесс обмена углеводов |
– |
Примечание: полужирным шрифтом выделен ген, внутри которого локализовано SNP
Дальнейшая аннотация включала в себя поиск источников литературы по генам, внутри которых локализованы SNP, с расчетом на дальнейшую разработку тест-систем.
Ген SLC2A13 в исследованиях B.B. Wang выявлен с помощью GWAS-анализа в программе GEMMA на коммерческих гибридах (пьетрен × дюрок) × (ландрас × йоркшир) и ассоциирован с содержанием внутримышечного жира [12]. При этом данный ген также был идентифицирован на свиньях породы ландрас при изучении полногеномного анализа по контрастным значениям конверсии корма [13]. Функциональная аннотация в программе DAVID позволила обнаружить его ассоциации с позитивной регуляцией формирования бета-амилоида – группы пептидов, образующихся из трансмембранного белка и состоящих из 40 аминокислотных остатков. Один из пептидов связывают с болезнью Альцгеймера у людей.
Уровень экспрессии гена NAV3 ассоциирован с глаукомой (прогрессирующая дегенерация зрительного нерва) у людей [14].
Ген CARMIL1 в исследованиях D. Shaoxiong et al. выявлен как специфичный для товарных помесей свиней (дюрок × (ландрас × йоркшир) и отвечающий за экстерьерный признак – обхват груди [15]. Он упоминается в исследованиях по гомозиготным участкам (ROH) свиней породы дюрок и связан со среднесуточным приростом [16].
Еще один ген – BMPR1B по анализу QTL отвечает за экстерьерные показатели, массу внутренних органов и воспроизводительные качества хряков и свиноматок разных пород свиней [17–19]. Аннотация по программе DAVID у данных генов не выявила биологических ассоциаций.
Экспрессия гена SNCA на опытных свиньях в исследованиях L. Knud et al. проводилась для сравнения белка с белком человека, так как дупликация и трипликация гена связана с болезнью Паркинсона [20].
Ген KATNAL1, как и ген TEX26 (сцепленный с участком выявленного SNP), напрямую взаимосвязан с одним из показателей кормового поведения свиней породы дюрок – временем, проведенным на автоматической кормовой станции [21]. По программе DAVID ген KATNAL1 ассоциирован со сперматогенезом и был обнаружен в исследованиях Y. Yang et al. [22] на популяции свиней Бама Сян (раннеспелая аборигенная китайская порода с черной шерстью), а также в исследованиях, посвященных изучению размера помета у свиней крупной белой породы [23].
Экспрессия гена MGAM2 связана с лучшей выживаемостью людей, у которых выявлен базальноподобный рак молочной железы, и ген коррелирует с иммунным ответом организма [24]. По программе DAVID аннотация связана с процессом обмена углеводов в организме свиней (Sus scrofa).
Заключение
Апробация данных на 800 головах свиней позволяет делать статистически обоснованные выводы как в отношении оценок племенной ценности, так и полногеномного анализа ассоциаций. Основой для проведенных исследований являлись значения оценок племенной ценности BLUP Animal Model и genomic BLUP по показателям эффективности использования корма и кормового поведения свиней породы дюрок. Так как конверсия корма – один из основных критериев эффективности производства в свиноводстве, для обнаружения его молекулярно-генетической детерминанты на полученных оценках был проведен полногеномный анализ ассоциаций, в результате которого идентифицированы SNP-маркеры и выявлены значимые гены – SLC2A13, CARMIL1, BMPR1B, KATNAL1.
Данные гены рекомендуется учитывать при оценке племенной ценности свиней породы дюрок. По выявленным генам, внутри которых локализованы SNP, будут разработаны и апробированы тест-системы для дальнейшего раннего прогнозирования племенной и геномной ценности свиней породы дюрок.
Исследования проведены при поддержке Министерства науки и высшего образования Российской Федерации. Регистрационный номер темы государственного задания FGGN-2022-0007
Литература
- Sanchez M.-P., Tribout T., Iannuccelli N., Bouffaud M., Servin B., Dehais, P. et al. A genome-wide association study of production traits in a commercial population of Large White pigs: Evidence of haplotypes affecting meat quality. Genet. Select. Evol., 2014. 46:12. DOI: 10.1186/1297-9686-46-12.
- Ding R., Yang M., Wang X., Quan J., Zhuang Z., Zhou S. et al. Genetic architecture of feeding behavior and feed efficiency in a Duroc pig population. Front. Genet., 2018. 9:220. DOI: 10.3389/fgene.2018.00220.
- Onteru S.K., Gorbach D.M., Young J.M., Garrick D.J., Dekkers J.C.M. and Rothschild M. F. Whole genome association studies of residual feed intake and related traits in the pig. PLoS One, 2013. 8:e61756. DOI: 10.1371/journal.pone.0061756.
- Silva E.F., Lopes M.S., Lopes P.S. and Gasparino E. A genome-wide association study for feed efficiency-related traits in a crossbred pig population. Animal, 2019. 13:2447–2456. DOI: 10.1017/s1751731119000910.
- Сермягин А.А., Нарышкина Е.Н., Карпушкина Т.В., Стрекозов Н.И., Зиновьева Н.А. Перспективы использования оценки геномной племенной ценности в селекции молочного скота. Молочное и мясное скотоводство, 2015. №7. С. 2–5.
- Сермягин А.А., Ермилов А.Н., Янчуков И.Н., Харитонов С.Н., Племяшов К.В., Тюренкова Е.Н., Стрекозов Н.И., Зиновьева Н.А. Региональная система геномной оценки как базовый элемент национальной программы генетического совершенствования крупного рогатого скота. Молочное и мясное скотоводство, 2017. №7. С. 3–7.
- Белоус А.А., Сермягин А.А., Костюнина О.В., Требунских Е.А., Зиновьева Н.А. Генетические и паратипические факторы, характеризующие эффективность использования корма у свиней породы дюрок. Сельскохозяйственная биология, 2018. Т. 53. №4. С. 712–722. DOI: 10.15389/agrobiology.2018.4.712rus.
- Белоус А.А., Требунских Е.А. Сравнительное исследование особенностей кормового поведения свиней пород ландрас и дюрок. Достижение науки и техники АПК, 2021. Т. 35. №10. С. 61–65. DOI: 10.53859/02352451_2021_35_10_61.
- Сермягин А.А., Гладырь Е.А., Харитонов С.Н., Ермилов А.Н., Стрекозов Н.И., Брем Г., Зиновьева Н.А. Полногеномный анализ ассоциаций с продуктивными и репродуктивными признаками у молочного скота в российской популяции голштинской породы. Сельскохозяйственная биология, 2016. Т. 51. №2. С. 182–193. DOI: 10.15389/agrobiology.2016.2.182rus.
- Белоус А.А., Сермягин А.А., Зиновьева Н.А. Генетическая оценка прогнозируемого остаточного потребления корма и экспрессия значимых генов-кандидатов свиней породы дюрок и товарных помесей второго поколения. Генетика, 2023. Т. 59. №11. С. 1253–1269. DOI: 10.31857/S0016675823110024.
- Отраднов П.И., Рудиянов Д.М., Белоус А.А. Валидация оценок племенной ценности свиней породы дюрок по признакам кормового поведения. Свиноводство, 2023. №5. С. 22–26. DOI: 10.37925/0039-713X-2023-5-22-26.
- Wang B.B., Hou L.M., Zhou W.D., Liu H., Tao W., Wu W.J., Niu P.P., Zhang Z.P., Zhou J., Li Q., Huang R.H., Li P.H. Genome-wide association study reveals a quantitative trait locus and two candidate genes on Sus scrofa chromosome 5 affecting intramuscular fat content in Suhuai pigs. Animal, 2021. 15(9):100341. DOI: 10.1016/j.animal.2021.100341.
- Tan Z., Wang Y., Yang T., Xing K., Ao H., Chen S., Zhang F., Zhao X., Liu J., Wang C. Differentially expressed genes in the caecal and colonic mucosa of Landrace finishing pigs with high and low food conversion ratios. Sci. Rep., 2017. 7(1):14886. DOI: 10.1038/s41598-017-14568-6.
- Chen J., Zhang C., Peng J., Tang C., Zhang C., Zhang M., Zou X., Zou Y. Gender-specific lncRNA-miRNA-mRNA regulatory network to reveal potential genes for primary open-angle glaucoma. Exp. Eye Res., 2023. 236:109668. DOI: 10.1016/j.exer.2023.109668.
- Deng S., Qiu Y., Zhuang Z., Wu J., Li X., Ruan D., Xu C., Zheng E., Yang M., Cai G., Yang J., Wu Z., Huang S. Genome-wide association study of body conformation traits in a three-way crossbred commercial pig population. Animals (Basel), 2023. 13(15):2414. DOI: 10.3390/ani13152414.
- Wang S., Yang J., Li G., Ding R., Zhuang Z., Ruan D., Wu J., Yang H., Zheng E., Cai G., Wang X., Wu Z. Identification of homozygous regions with adverse effects on the five economic traits of Duroc pigs. Front. Vet. Sci., 2022. 9:855933. DOI: 10.3389/fvets.2022.855933.
- Fan B., Onteru S.K., Mote B.E., Serenius T., Stalder K.J., Rothschild M.F. Large-scale association study for structural soundness and leg locomotion traits in the pig. Genet. Sel. Evol., 2009. 41(1):14. DOI: 10.1186/1297-9686-41-14.
- Tremoen N.H., Van Son M., Andersen-Ranberg I., Grindflek E., Myromslien F.D., Gaustad A.H., Våge D.I. Association between single-nucleotide polymorphisms within candidate genes and fertility in Landrace and Duroc pigs. Acta Vet. Scand., 2019. 61(1):58. DOI: 10.1186/s13028-019-0493-x.
- Li X., Wu J., Zhuang Z., Ye Y., Zhou S., Qiu Y., Ruan D., Wang S., Yang J., Wu Z., Cai G., Zheng E. Integrated single-trait and multi-trait GWASs reveal the genetic architecture of internal organ weight in pigs. Animals (Basel), 2023. 13(5):808. DOI: 10.3390/ani13050808.
- Larsen K., Bæk R., Sahin C, Kjær L., Christiansen G., Nielsen J., Farajzadeh L., Otzen D.E. Molecular characteristics of porcine alpha-synuclein splicing variants. Biochimie, 2021. 180:121–133. DOI: 10.1016/j.biochi.2020.10.019.
- Do D.N., Strathe A.B., Ostersen T., Jensen J., Mark T., Kadarmideen H.N. Genome-wide association study reveals genetic architecture of eating behavior in pigs and its implications for humans obesity by comparative mapping. PLoS One, 2013. 8(8):e71509. DOI: 10.1371/journal.pone.0071509.
- Yang Y., Adeola A.C., Xie H.B., Zhang Y.P. Genomic and transcriptomic analyses reveal selection of genes for puberty in Bama Xiang pigs. Zool. Res., 2018. 39(6):424–430. DOI: 10.24272/j.issn.2095-8137.2018.068.
- Sell-Kubiak E., Knol E.F., Lopes M. Evaluation of the phenotypic and genomic background of variability based on litter size of Large White pigs. Genet. Sel. Evol., 2022. 54(1):1. DOI: 10.1186/s12711-021-00692-5.
- Xu S., Feng Y., Zhao S. Proteins with evolutionarily hypervariable domains are associated with immune response and better survival of basal-like breast cancer patients. Comput. Struct. Biotechnol. J., 2019. 17:430–440. DOI: 10.1016/j.csbj.2019.03.008.