В 2025 году журналу
ИСПОЛНИТСЯ
95 ЛЕТ!
ЧИТАЙТЕ
Материалы к юбилею >>>

РУБРИКИ
 Племенное дело |
 Крупный план |
 Актуально |
 Корма |
 Техническая политика |
 Событие |
 Ветеринария |
 Эксклюзивное интервью |
 Статистика |
 Выставки |
Творчество
наших читателей
ЧИТАЙТЕ
Автор С.И. КАЛИНИЧЕНКО
ГИМН СВИНЬЕ >>>
СТАТЬИ ИЗ НОМЕРА
DOI: 10.37925/0039-713X-2024-5-21-25
УДК 636.475
ДИНАМИКА АЛЛЕЛОФОНДА ЛИВЕНСКОЙ ПОРОДЫ СВИНЕЙ НА ОСНОВЕ МИКРОСАТЕЛЛИТНОГО АНАЛИЗА
Т.Е. ДЕНИСКОВА, кандидат биолог. наук, вед. научный сотрудник, А.С. АБДЕЛЬМАНОВА, доктор биолог. наук, ст. научный сотрудник, В.Р. ХАРЗИНОВА, кандидат биолог. наук, вед. научный сотрудник, Н.А. ЗИНОВЬЕВА, доктор биолог. наук, профессор, академик РАН, директор, ФГБНУ ФИЦ ВИЖ имени Л.К. Эрнста
Проведена оценка аллелофонда популяций ливенской породы в пятилетней динамике (с 2018 по 2022 год) с использованием мультилокусной панели микросателлитов в сравнении с коммерческими породами (крупная белая и ландрас). Выявлены аллели, характерные для ливенской породы и отсутствующие у коммерческих пород. Тем не менее, отмечена тенденция к утрате определенных аллелей в динамике анализируемого отрезка времени. Установлено, что популяция ливенской породы 2022 года по уровню наблюдаемой гетерозиготности достоверно превосходила (P<0,05) коммерческие породы. На основе анализа главных компонент, построения генетической сети и кластерного анализа показано сохранение аутентичных геномных компонентов, свойственных ливенской породе, в современной популяции.
Ключевые слова: STR-маркеры, локальные породы, генетическое разнообразие, аллельный профиль.
Dynamics of the allelepool of the Livni pig breed based on microsatellite analysis
T.E. DENISKOVA, candidate of biological sciences, leading researcher, A.S. ABDELMANOVA, doctor of biological sciences, senior researcher, V.R. KHARZINOVA, candidate of biological sciences, leading researcher, N.A. ZINOVIEVA, doctor of biological sciences, professor, academician of the RAS, director, L.K. Ernst Federal Research Center for Animal Husbandry
Using a multilocus panel of microsatellites, the allele pool of Livni breed populations was assessed in five-year dynamics (from 2018 to 2022) and in comparison with commercial breeds (Large White and Landrace). We identified alleles, which were found in Livni breed and absent in commercial breeds. However, there was a tendency towards the loss of certain alleles in the dynamics of the analyzed period. It was shown that observed heterozygosity in the population of Livni breed in 2022 was significantly higher (P<0.05) compared to those estimated in commercial breeds. Based on principal component analysis, genetic network construction, and cluster analysis, the preservation of authentic genomic components, which were predominant in Livni breed, in the modern population is shown.
Key words: STR markers, local breeds, genetic diversity, allelic profile.
Введение
В структуре российского свиноводства преобладают высокопродуктивные коммерческие породы, тогда как незначительная доля приходится на отечественные породы (С.В. Павлова с соавт., 2021). По мясным и откормочным качествам отечественные породы уступают зарубежным.
Однако отечественные породы свиней, среди которых особенно выделяется ливенская порода, обладают высокими адаптационными качествами и неприхотливостью к условиям кормления и содержания.
Ливенская порода создана под руководством Н.Н. Коровецкой в Орловской области путем скрещивания аборигенных вислоухих свиней и хряков крупной белой, беркширской и польско-китайских пород (Л.К. Эрнст с соавт., 1994). По мнению В.С. Никульникова с соавт. (2016), ливенская порода – это лучшая ресурсосберегающая отечественная порода свиней, демонстрирующая хорошие откормочные качества при кормлении небольшим количеством концентрированных кормов.
В последние годы из-за специфики технологии разведения и рыночного спроса локальные породы сельскохозяйственных животных подвергаются генетической эрозии или уже находятся под риском исчезновения (L. Temerario et al., 2023). С целью защиты генетического разнообразия мониторинг с использованием молекулярно-генетических подходов имеет большое значение для оценки траекторий развития популяций и поддержки реализации превентивных программ, направленных на сохранение важных и отдельных локальных популяций (M.W. Bruford et al., 2017).
В течение последних 20–30 лет микросателлиты (STR-маркеры) были предпочтительными ДНК-маркерами для проведения популяционно-генетических исследований сельскохозяйственных животных. Однако в последние годы с развитием технологий проведения высокопроизводительного генотипирования, в частности разработки коммерческих ДНК-чипов, однонуклеотидные полиморфизмы (SNP) приобретают все большую популярность.
Намечается следующая тенденция: столкнувшись с мощью ДНК-чипов, микросателлиты все чаще остаются в стороне в исследованиях, посвященных геномному разнообразию (A. Laoun et al., 2020). Тем не менее продемонстрировано, что популяционная структура и генетические дифференциации, оцененные с помощью микросателлитов, соответствуют паттерну, полученному с использованием ДНК-чипов (A. Laoun et al., 2020). Таким образом, микросателлитный анализ – это надежный и доступный молекулярно-генетический инструмент, который позволяет быстро оценить изменения аллелофонда в популяциях сельскохозяйственных животных.
За последние пять лет с помощью микросателлитов, рекомендованных ISAG-FAO, был решен довольно широкий спектр генетических задач в свиноводстве. Так, например, на основе применения панели из 21 микросателлита впервые была оценена генетическая структура разнообразных популяций филиппинских аборигенных свиней, принадлежащих к восточной, юго-восточной, тихоокеанской и кордильерской кладам (J.B. Banayo et al., 2023) На основе полученных результатов были определены приоритетные группы для разработки программ по сохранению этих эндемичных генетических ресурсов (J.B. Banayo et al., 2023).
Мультилокусная панель, состоящая из 13 микросателлитов, была успешно использована для установления структуры популяции креольских свиней, которые в силу своей неприхотливости представляют собой источник экономического дохода в сельских общинах в Перу (R. Luna et al., 2023). Изучены генетические взаимосвязи вьетнамских аборигенных свиней (N.V. Ba et al., 2020). 29 микросателлитов выявили четкую географическую дивергенцию у групп тайских аборигенных свиней, обитающих в трех географических областях (K. Chaweewan et al., 2023). Кроме того, установлено, что тайские свиньи значительно отличаются от европейских и китайских пород и имеют признаки интрогрессии с диким кабаном, что может привести к генетической эрозии.
Исследование аллелофонда восьми локальных пород свиней России, включая ливенскую, и трех коммерческих пород с использованием микросателлитов показало, что локальные породы превосходили группы свиней зарубежного происхождения по аллельному и генетическому разнообразию (В.Р. Харзинова, Н.А. Зиновьева, 2020). Однако понимание процессов, происходящих в аллелофонде популяций в динамике нескольких поколений, существенно для внесения своевременных корректировок в подбор родительских пар (например, для предотвращения нарастания инбридинга). Так, в частности, анализ полиморфизма 11 микросателлитов в популяциях овец романовской породы, проведенный в динамике трех лет, показал увеличение количества гетерозигот (Т.Е. Денискова с соавт., 2017).
Цель работы – проанализировать состояние аллелофонда популяций ливенской породы в течение пяти лет (с 2018 по 2022 год) и оценить дифференциацию этой локальной группы свиней от наиболее популярных коммерческих пород (крупная белая и ландрас).
Материалы и методы
Исследования проводили на базе ЦКП «Биоресурсы и биоинженерия сельскохозяйственных животных» ФГБНУ ФИЦ ВИЖ имени Л.К. Эрнста. Материалом для исследований служили образцы биологического материала (ушной выщип) свиней ливенской породы разных лет. Образцы ландраса и крупной белой породы были добавлены в выборку в качестве групп сравнения, поскольку указанные породы используются при разведении ливенской (табл. 1). Биологический материал пород, задействованных в исследовании, депонирован в генетическом банке ФГБНУ ФИЦ ВИЖ имени Л.К. Эрнста.
Таблица 1. Характеристика исследуемых популяций.
|
Порода |
Год отбора образцов |
Сокращение |
Кол-во образцов |
|
Ливенская |
2018 |
LIV_2018 |
66 |
|
Ливенская |
2019 |
LIV_2019 |
242 |
|
Ливенская |
2020 |
LIV_2020 |
218 |
|
Ливенская |
2021 |
LIV_2021 |
121 |
|
Ливенская |
2022 |
LIV_2022 |
172 |
|
Крупная белая |
2023 |
LW |
100 |
|
Ландрас |
2023 |
LN |
100 |
Генетическое разнообразие оценивали с использованием панели девяти высокополиморфных STR-маркеров, рекомендованных ISAG, – S0155, S0005, SW72, SW951, S0386, S0355, SW240, SW857, S0101. Геномную ДНК выделяли с помощью наборов «ДНК-Экстран-2» (ЗАО «Синтол», Россия) в соответствии с инструкциями производителя. Для исследования микросателлитов применяли метод полимеразной цепной реакции (ПЦР). Анализ продуктов амплификации проводили методом капиллярного электрофореза с использованием ДНК-анализатора 3130xl Genetic Analyzer (Applied Biosystems, США) с лазерной детекцией в среде полимера РОР-6. Определение длин амплифицированных фрагментов производили посредством программы GeneMapper® с использованием в качестве фрагментного стандарта GeneScan™-350 ET ROX.
Статистическую обработку и визуализацию данных проводили в программной среде R 3.5.0 (R Core Team) с использованием дополнительных R-пакетов. Вероятность идентичности генотипов (probability of identity, PI) и вероятность исключения родителей (probability of exclusion, PE) рассчитывали в программе GenALEx (R. Peakall, P.E. Smouse, 2012). Степень генетической дифференциации популяций оценивали в программе Structure 2.3.4 (J.K. Pritchard et al., 2000). Оптимальное число К определяли с помощью программы Structure Harvester (D.A. Earl, B.M. VonHoldt, 2012).
Результаты исследований
Анализ генетического разнообразия показал, что наибольшим уровнем наблюдаемой (Ho) гетерозиготности характеризовалась популяция ливенской породы 2022 года, достоверно (P<0,05) превышая по этому показателю обе коммерческие породы и популяцию ливенской породы 2021 года. Аналогичные тенденции были обнаружены при сравнении популяций ливенской породы 2008 и 2018 годов (М.М. Сомова с соавт., 2019).
Аллельное разнообразие было максимальным в популяции ливенской породы 2019 года, после чего в породе началось некоторое уменьшение аллельного разнообразия, достигнувшее в 2021 году минимума, сопоставимого с коммерческими породами. Некоторое повышение аллельного разнообразия в популяции 2022 года, возможно, свидетельствует об обновлении производящего состава. В целом значения генетического и аллельного разнообразия ливенской породы сходны со значениями, рассчитанными для других отечественных пород свиней (В.Р. Харзинова, Н.А. Зиновьева, 2020).
Поскольку 95%-ный доверительный интервал (ДИ) коэффициента инбридинга (uFis) располагается в диапазоне как положительных, так и отрицательных чисел, переходя через ноль, мы не можем считать значения данного показателя статистически значимыми (табл. 2).
Таблица 2. Характеристика генетического разнообразия популяций ливенской породы разных лет
|
Популяция |
n |
Ho |
uHe |
Ar |
uFis [ДИ 95%] |
|
LIV_2018 |
66 |
0,673±0,036 |
0,673±0,041 |
6,111±0,978 |
-0,017 [-0,124; 0,09] |
|
LIV_2019 |
242 |
0,648±0,043 |
0,700±0,036 |
6,549±1,183 |
0,070 [-0,011; 0,151] |
|
LIV_2020 |
218 |
0,646±0,054 |
0,681±0,039 |
6,287±1,201 |
0,048 [-0,066; 0,162] |
|
LIV_2021 |
121 |
0,538±0,082 |
0,593±0,059 |
5,887±1,143 |
0,146 [-0,058; 0,35] |
|
LIV_2022 |
172 |
0,713±0,034 |
0,685±0,035 |
6,288±1,156 |
-0,044 [-0,109; 0,021] |
|
LW |
100 |
0,581±0,077 |
0,589±0,073 |
5,176±1,033 |
0,008 [-0,063; 0,079] |
|
LN |
100 |
0,539±0,085 |
0,529±0,085 |
5,043±1,051 |
-0,024 [-0,063; 0,015] |
Примечание. Здесь и далее: LIV_ – популяции ливенской породы соответствующего года, LW – крупная белая порода, LN – ландрас, n – количество образцов, Ho – наблюдаемая гетерозиготность, uHe – несмещенная ожидаемая гетерозиготность, Ar – аллельное разнообразие, uFis – несмещенный коэффициент инбридинга (доверительный интервал 95%).
Вероятность идентичности генотипов (PI) у ливенской породы всех групп, кроме популяции 2021 года, была ниже, чем для обеих коммерческих пород, что свидетельствует о большем аллельном разнообразии, присущем локальной породе, сохранявшемся на протяжении ряда лет. Наиболее сильные различия заметны уже при комбинации из пяти локусов, когда аллельное разнообразие в популяциях ливенской породы (кроме группы 2021 года) отличается в десятки раз.
Наименьшим аллельным разнообразием отличается ландрас, для которого вероятность идентичности генотипов двух случайных особей в выборке при анализе по девяти микросателлитным локусам составляет 1,2*10-6. При этом у ливенской породы вероятность идентичности варьировала от 2,2*10-7 в популяции 2021 года до 6,0*10-9 в популяции 2019 года (табл. 3).
Таблица 3. Вероятность идентичности генотипов особей в исследованных популяциях при анализе по разному количеству локусов
|
Популяция |
n |
Кол-во локусов |
||||||||
|
1 |
2 |
3 |
4 |
5 |
6 |
7 |
8 |
9 |
||
|
LIV_2018 |
66 |
1,9*10-1 |
5,5*10-3 |
1,2*10-3 |
2,9*10-4 |
4,7*10-5 |
4,6*10-6 |
6,8*10-7 |
5,7*10-8 |
1,9*10-8 |
|
LIV_2019 |
242 |
1,6*10-1 |
4,2*10-3 |
1,1*10-3 |
2,5*10-4 |
4,0*10-5 |
3,0*10-6 |
4,4*10-7 |
2,9*10-8 |
6,0*10-9 |
|
LIV_2020 |
218 |
1,8*10-1 |
3,3*10-3 |
8,5*10-4 |
2,0*10-4 |
3,9*10-5 |
6,0*10-6 |
8,6*10-7 |
4,7*10-8 |
1,1*10-8 |
|
LIV_2021 |
121 |
2,0*10-1 |
5,0*10-3 |
1,8*10-3 |
4,1*10-4 |
1,1*10-4 |
5,8*10-5 |
1,4*10-5 |
9,8*10-7 |
2,2*10-7 |
|
LIV_2022 |
172 |
1,5*10-1 |
2,7*10-3 |
5,3*10-4 |
1,3*10-4 |
2,2*10-5 |
3,8*10-6 |
7,6*10-7 |
5,3*10-8 |
1,0*10-8 |
|
LW |
100 |
3,2*10-1 |
1,6*10-2 |
2,5*10-3 |
8,7*10-4 |
2,1*10-4 |
5,4*10-5 |
4,2*10-5 |
3,0*10-6 |
3,4*10-7 |
|
LN |
100 |
2,0*10-1 |
4,6*10-3 |
1,6*10-3 |
9,6*10-4 |
3,3*10-4 |
1,3*10-4 |
1,0*10-4 |
6,6*10-6 |
1,2*10-6 |
Вероятность исключения родителей (PE) для всех групп ливенской породы, кроме популяции 2021 года, составляет более 0,999 уже при комбинации из шести локусов. В популяциях крупной белой и ливенской 2021 года для уверенной идентификации животного (более 0,999) необходимо использовать комбинацию минимум из восьми локусов, что подтверждает меньшее аллельное разнообразие этих групп. У ландраса требуется применять комбинацию из девяти генотипированных локусов, чтобы уверенно (с вероятностью более 0,999) исключить родство животных, что свидетельствует о наименьшем количестве разнообразных аллелей в данной породе (табл. 4).
Таблица 4. Вероятность идентификации животного в исследованных популяциях при анализе по разному количеству локусов
|
Популяция |
n |
Кол-во локусов |
||||||||
|
1 |
2 |
3 |
4 |
5 |
6 |
7 |
8 |
9 |
||
|
LIV_2018 |
66 |
0,5569 |
0,9562 |
0,9783 |
0,9886 |
0,9957 |
0,9988 |
0,9995 |
0,9999 |
0,9999 |
|
LIV_2019 |
242 |
0,5994 |
0,9648 |
0,9807 |
0,9905 |
0,9963 |
0,9991 |
0,9997 |
0,9999 |
1,0000 |
|
LIV_2020 |
218 |
0,5665 |
0,9727 |
0,9848 |
0,9925 |
0,9966 |
0,9987 |
0,9995 |
0,9999 |
1,0000 |
|
LIV_2021 |
121 |
0,5336 |
0,9606 |
0,9751 |
0,9873 |
0,9933 |
0,9948 |
0,9973 |
0,9995 |
0,9997 |
|
LIV_2022 |
172 |
0,6184 |
0,9764 |
0,9888 |
0,9943 |
0,9977 |
0,9990 |
0,9996 |
0,9999 |
1,0000 |
|
LW |
100 |
0,3637 |
0,9023 |
0,9589 |
0,9756 |
0,9878 |
0,9932 |
0,9939 |
0,9987 |
0,9996 |
|
LN |
100 |
0,4997 |
0,9609 |
0,9742 |
0,9789 |
0,9868 |
0,9909 |
0,9917 |
0,9984 |
0,9992 |
Проведен детальный анализ аллельного разнообразия для исследованных групп и выявлены аллели, характерные для ливенской породы и в то же время отсутствующие как у крупной белой, так и у ландраса (табл. 5).
Таблица 5. Частоты аллелей, характерные для ливенской породы
|
Локус |
Аллель |
Популяция |
||||
|
LIV_2018 |
LIV_2019 |
LIV_2020 |
LIV_2021 |
LIV_2022 |
||
|
S0155 |
154 |
0,053 |
0,054 |
0,037 |
0,029 |
0,047 |
|
S0155 |
156 |
0,000 |
0,000 |
0,000 |
0,004 |
0,000 |
|
S0155 |
162 |
0,038 |
0,083 |
0,064 |
0,045 |
0,087 |
|
S0005 |
225 |
0,000 |
0,019 |
0,096 |
0,025 |
0,029 |
|
S0005 |
227 |
0,000 |
0,002 |
0,028 |
0,000 |
0,000 |
|
S0005 |
229 |
0,000 |
0,019 |
0,002 |
0,004 |
0,006 |
|
S0386 |
170 |
0,000 |
0,021 |
0,023 |
0,004 |
0,017 |
|
S0386 |
180 |
0,053 |
0,000 |
0,000 |
0,000 |
0,000 |
|
S0386 |
182 |
0,000 |
0,066 |
0,000 |
0,000 |
0,052 |
|
S0355 |
253 |
0,008 |
0,019 |
0,000 |
0,000 |
0,000 |
|
S0355 |
255 |
0,008 |
0,000 |
0,009 |
0,000 |
0,000 |
|
S0355 |
265 |
0,106 |
0,083 |
0,032 |
0,000 |
0,038 |
Как видно из таблицы 5, в локусе S0155 аллели 154 и 162 встречаются у всех популяций ливенской породы с разной частотой. Некоторые аллели (локус S0386, аллель 180; локус S0355, аллель 255; локус S0355, аллель 265), обнаруженные в более ранних группах, были утрачены в популяциях последующих лет.
С целью сравнения изменений частот встречаемости аллелей были составлены аллельные профили для каждой исследуемой группы. Для каждого локуса в отдельности были определены аллели с максимальной частотой встречаемости (рис. 1). Такие аллели были приняты в качестве характерных (наиболее часто встречающихся) для данной популяции.
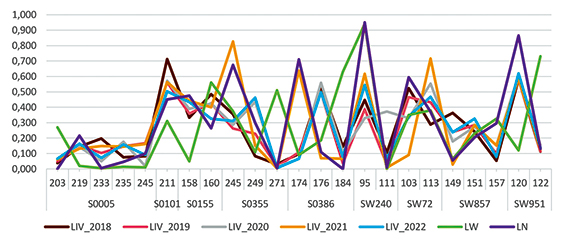
Рис. 1. Аллельные профили свиней исследованных пород на основании анализа микросателлитных локусов
Как видно из рисунка 1, наиболее сильно породы отличались по локусу S0005, в нем идентифицировано пять аллелей, разных для исследованных групп, только аллель 219 был общим для ливенской популяции 2022 года и ландраса.
В локусе S0101 наиболее часто встречался аллель 211, общий для всех пород.
Дальнейшее изучение представленных профилей выявило следующие тенденции: ландрас и популяция ливенской породы 2021 года были схожи между собой по встречаемости одинаковых аллелей (шесть из девяти, а именно: 211 в S0101; 158 в S0155; 245 в S0355; 174 в S0386; 95 в SW240; 120 в SW951). Крупная белая порода показала одинаковые аллели с ливенскими популяциями 2018, 2019 и 2020 годов по двум локусам (211 в S0101; 160 в S0155).
Проведен анализ взаимоотношений между исследованными популяциями. Анализ главных компонент показал, что большинство образцов ливенской породы разных лет образуют перекрывающееся облако (рис. 2). При этом образцы 2021 года несколько отделяются от общего массива и расположены ближе к кластеру, образуемому животными породы ландрас, что может говорить об их большей генетической близости. Тем не менее стоит отметить, что животные ливенской породы сохраняют достаточно четкую дифференциацию от коммерческих пород в течение ряда лет, что свидетельствует о консолидированности их генофонда.
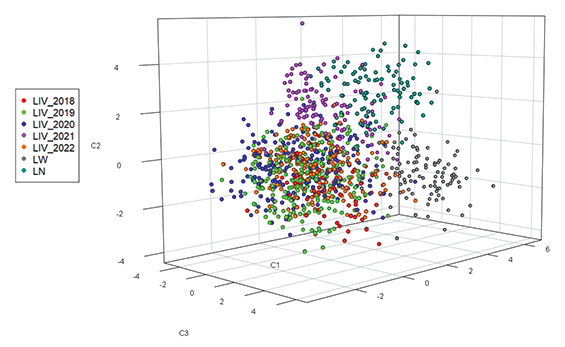
Рис. 2. Генетическая дифференциация исследованных популяций на основании результатов анализа главных компонент
Анализ генетической дифференциации популяций с помощью программы Structure 2.3.4 показал, что при К=2 коммерческие породы (ландрас и крупная белая) и популяция ливенской породы 2021 года отделяются от остальных популяций ливенской породы (рис. 3). При К=3 сохраняется обособленность коммерческих пород, а в группе ливенской породы выделяется дополнительный кластер, включающий популяции 2020, 2021 и частично 2022 года. При этом в группе 2021 года присутствуют генетические компоненты коммерческих пород, а в группе 2022 года наблюдается примесь генетических компонентов более ранних популяций ливенской породы (2018 и 2019 годов).
При увеличении числа кластеров до К=4 коммерческие породы образуют породоспецифичные кластеры, в группе ливенской породы 2021 года наблюдается значительная доля примеси компонентов, свойственных ландрасу. Популяции ливенской породы 2018 и 2019 годов большей частью несут общие генетические компоненты, сохранившиеся в популяции 2022 года. Популяция ливенской породы 2020 года сформирована животными, отличающимися от представителей прошлых лет, что может быть связано со сменой производящего состава и (или) изменением направления селекционной работы. В пользу этой гипотезы говорит практически полное поглощение ландрасом в образцах ливенской породы 2021 года. Однако в 2022 году отмечается некоторое восстановление доли аутентичных компонентов ливенской породы.
Интересно, что мы не нашли какого-либо документального подтверждения привлечения генетического материала хряков породы ландрас для улучшения продуктивных качеств ливенской породы. Тем не менее, в литературе имеются сообщения, что другие коммерческие породы были использованы с этой целью. Так, например, известно, что скрещивание свиноматок ливенской породы с хряками крупной белой породы повышало сохранность поросят-сосунов и живую массу гнезда в двухмесячном возрасте (А.И. Дедкова, Н.Н. Сергеева, 2011). Однако данные этого исследования не подтвердили наличие генетических компонентов, свойственных крупной белой породе, во всех изученных популяциях свиней ливенской породы. Кроме того, по информации О.А. Михайловой (2019), производились пробные скрещивания свиноматок ливенской породы с хряками породы пьетрен. В такой выборке отсутствовали микросателлитные профили породы пьетрен, что не дает возможности провести анализ вероятного остаточного влияния этой породы на генофонд современной популяции ливенской породы.
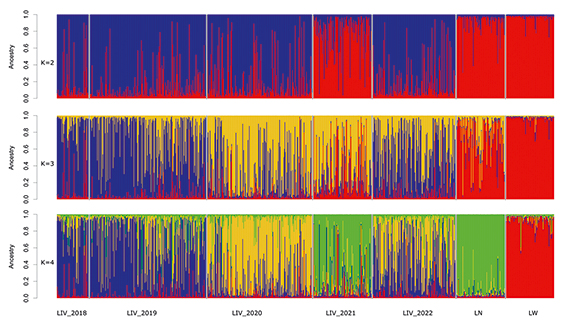
Рис. 3. Характеристика генетической структуры ливенской породы разных лет в сравнении с коммерческими породами свиней
На дендрограмме, построенной на основании значений FST, видно, что популяции ливенской породы образуют отдельные группы (рис. 4). При этом LIV_2018 и LIV_2020 находятся на некотором удалении от общей грани. Популяция LIV_2021 удалена от основной группы ливенской породы, располагаясь вблизи ландраса, не образует с ним единой ветви, что свидетельствует о некоторой генетической обособленности от этой породы.
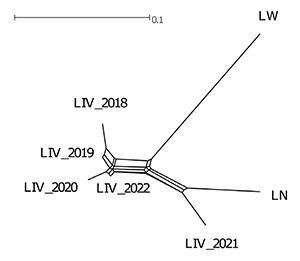
Рис. 4. Дендрограмма генетических дистанций между исследованными популяциями свиней
Заключение
Проведенные исследования показали, что в настоящее время ливенская порода свиней еще является уникальной и обособленной от коммерческих пород, несмотря на использование их в качестве улучшающих. Тем не менее наблюдается постепенная утрата аллельных комбинаций, свойственных более ранним популяциям ливенской породы. Аллельный профиль постепенно меняется, приобретая очертания, характерные для коммерческих пород.
С целью дальнейшего использования ливенской породы как источника уникальных аллельных сочетаний в программах устойчивого развития свиноводства необходима разработка селекционных программ, направленных не только на улучшение продуктивных качеств животных, но и на поддержание биоразнообразия и самобытности ливенской породы.
Работа выполнена при поддержке Российского научного фонда, проект №23-46-00014
Литература
- Дедкова А.И., Сергеева Н.Н. Улучшение продуктивных качеств свиней ливенской породы путем применения двухпородного скрещивания. Вестник аграрной науки, 2011. №6(33). С. 32–34.
- Денискова Т.Е., Соловьева А.Д., Костюнина О.В., Зиновьева Н.А. Динамика аллелофонда овец романовской породы на основании анализа микросателлитов. Овцы, козы, шерстяное дело, 2017. №3. С. 5–6.
- Михайлова О.А. Совершенствование мясной продуктивности свиней ливенской породы в условиях крестьянско-фермерских хозяйств. Вестник аграрной науки, 2019. №4(79). С. 65–72. http://dx.doi.org/10.15217/48484.
- Никульников В.С., Овсянникова Н.Н., Ляхова О.Л., Сысоева Л.А., Ефанов А.М. Ливенская порода свиней – ценный генофонд России. Актуальные проблемы естественнонаучного образования, защиты окружающей среды и здоровья человека, 2016. №4(4). С. 251–255.
- Павлова С.В., Козлова Н.А., Мышкина М.С., Щавликова Т.Н. Генетический потенциал племенного свиноводства в настоящее время. Эффективное животноводство, 2021. № 9(175). С. 81–83.
- Сомова М.М., Харзинова В.Р., Сермягин А.А., Костюнина О.В., Мельникова Е.Е., Шеламова Н.Н., Алдобаев А.М., Зиновьева Н.А. Обоснованность сохранения ливенской породы свиней с точки зрения уникальности генофонда современной популяции. Зоотехния, 2019. №10. С. 10–15. DOI: 10.25708/ZT.2019.71.17.003.
- Харзинова В.Р., Зиновьева Н.А. Паттерн генетического разнообразия у локальных и коммерческих пород свиней на основе анализа микросателлитов. Вавиловский журнал генетики и селекции, 2020. №24(7). С. 747–754. DOI: 10.18699/VJ20.669.
- Эрнст Л.К. Генетические ресурсы сельскохозяйственных животных в России и сопредельных странах/Л. К. Эрнст и др. СПб: Всероссийский НИИ генетики и разведения с.-х. животных, 1994. 469 с.
- Ba N.V., Arakawa A., Ishihara S., Nam L.Q., Thuy T.T.T., Dinh N.C., Ninh P.H., Cuc N.T.K., Kikuchi K., Pham L.D., Taniguchi M. Evaluation of genetic richness among Vietnamese native pig breeds using microsatellite markers. Anim. Sci. J., 2020. №91(1). P. 13343. DOI: 10.1111/asj.13343.
- Banayo J.B., Manese K.L.V., Furusho K.O., Salces A.J., Yamagata T. Genetic diversity and population structure analysis of Philippine native pigs highlight five priority populations for conservation. Ecol. Evol., 2023. №13(11). P. e10618. DOI: 10.1002/ece3.10618.
- Bruford M.W. Monitoring changes in genetic diversity/M.W. Bruford, N. Davies, M.E. Dull, D.P. Faith, M. Walters. Springer, 2017. DOI: 10.1007/978-3-319-27288-7_5.
- Chaweewan K., Mahinchai P., Kongsook S., Soponchit S., Weerasamith P., Awiruttapanich W., Prapawat P., Jamparat W., Chanthaworn T., Rattanamahavichai N., Weangchanok S., Arikit S., Duangjinda M., Tuntivisoottikul K., Chaosap C., Jirajaroenrat K. Genetic divergence of Thai indigenous pigs from three distinct geographic regions revealed by microsatellite marker analysis. Animals (Basel), 2023. №13(4). P. 625. DOI: 10.3390/ani13040625.
- Earl D.A., VonHoldt B.M. Structure harvester: A website and program for visualizing structure output and implementing the Evanno method. Conservation Genetics Resources, 2012. №4(2). P. 359–361. DOI: 10.1007/s12686-011-9548-7.
- Laoun A., Harkat S., Lafri M., Gaouar S.B.S., Belabdi I., Ciani E., De Groot M., Blanquet V., Leroy G., Rognon X. et al. Inference of breed structure in farm animals: Empirical comparison between SNP and microsatellite performance. Genes, 2020. №11(1). P. 57. https://doi.org/10.3390/genes11010057.
- Luna R., Acuña W., Gutiérrez G., Castro Muñoz M.D.R., Veli E. Genetic diversity in creole pigs in south central Peru. Trop. Anim. Health Prod., 2023. №55(6). P. 426. DOI: 10.1007/s11250-023-03839-2.
- Peakall R., Smouse P.E. GenAlEx 6.5: Genetic analysis in Excel. Population genetic software for teaching and research-an update. Bioinformatics, 2012. №28. P. 2537–2539.
- Pritchard J.K., Stephens M., Donnelly P. Inference of population structure using multilocus genotype data. Genetics, 2000. №155. P. 945–959.
- R Core Team. R: A language and environment for statistical computing. R Foundation for statistical computing. Austria, Vienna, 2012. http://www.Rproject.org.
- Temerario L., Monaco D., Mastrorocco A., Martino N.A., Cseh S., Lacalandra G.M., Ciani E., Dell’Aquila M.E. New strategies for conservation of Gentile di Puglia sheep breed, an autochthonous capital of millennial tradition in southern Italy. Animals, 2023. №13(14). P. 2371. https://doi.org/10.3390/ani13142371.








