В 2025 году журналу
ИСПОЛНИТСЯ
95 ЛЕТ!
ЧИТАЙТЕ
Материалы к юбилею >>>

РУБРИКИ
 Племенное дело |
 Крупный план |
 Актуально |
 Корма |
 Техническая политика |
 Событие |
 Ветеринария |
 Эксклюзивное интервью |
 Статистика |
 Выставки |
Творчество
наших читателей
ЧИТАЙТЕ
Автор С.И. КАЛИНИЧЕНКО
ГИМН СВИНЬЕ >>>
СТАТЬИ ИЗ НОМЕРА
DOI: 10.37925/0039-713X-2024-4-8-13
УДК 636.2.033
ГЕНЕТИЧЕСКАЯ АРХИТЕКТУРА ВОСПРОИЗВОДИТЕЛЬНЫХ КАЧЕСТВ СВИНОМАТОК КРУПНОЙ БЕЛОЙ ПОРОДЫ
А.А. БЕЛОУС1, кандидат биолог. наук, ст. научный сотрудник, e-mail: belousa663@gmail.com, П.И. ОТРАДНОВ1, мл. научный сотрудник, Е.А. ТРЕБУНСКИХ2, аспирант, зам. директора по племенному делу, А.Ф. КОНТЭ1, кандидат с.-х. наук, ст. научный сотрудник, А.А. РЕШЕТНИКОВА1, аспирант, мл. научный сотрудник, В.В. ВОЛКОВА1, кандидат биолог. наук, ст. научный сотрудник, 1ФГБНУ ФИЦ ВИЖ имени Л.К. Эрнста, 2селекционно-гибридный центр «Топ Ген»
Основной целью данного исследования было определение генетических параметров репродуктивных признаков популяции свиней крупной белой породы, в частности веса поросят при рождении, веса поросят при отъеме, числа живорожденных и мертворожденных поросят, мумифицированного помета. Выявлено 162 достоверных SNP, из которых шесть были общими для ряда признаков. Функциональная аннотация по биологическим функциям (GO) установила 12 значимых генов-кандидатов, среди которых особое внимание следует обратить на ген ADCY5, ранее демонстрировавший влияние на репродуктивные способности других видов сельскохозяйственных животных и птицы.
Ключевые слова: полногеномное ассоциативное исследование, однонуклеотидные полиморфизмы, воспроизводительные признаки, свиноматки крупной белой породы.
Genetic architecture of reproductive performance of sows of the Large White breed
A.A. BELOUS1, candidate of biological sciences, senior researcher, P.I. OTRADNOV1, junior researcher, E.A. TREBUNSKIKH2, post-graduate student, Deputy Director for breeding, A.F. CONTE1, candidate of agricultural sciences, senior researcher, A.A. RESHETNIKOVA1, junior researcher, V.V. VOLKOVA1, candidate of biological sciences, senior researcher, 1Federal Research Center for Animal Husbandry named after Academy Member L.K. Ernst, 2Selection and Hybrid Center Top Gen
The main goal of this study was to determine the genetic parameters of reproductive traits in a population of Large White pigs, in particular, the birth weight of piglets, weaning weight of piglets, number of live-born and stillborn piglets, and mummified litter. We identified 162 significant SNPs, of which 6 were common for a number of traits. Functional annotation by biological functions (GO) identified 12 significant candidate genes, among which special attention should be paid to the ADCY5 gene, which has previously demonstrated an influence on reproductive abilities in other agricultural animal and poultry species.
Key words: genome-wide association study, single nucleotide polymorphisms, reproductive traits, Large White sows.
Введение
В свиноводстве репродуктивные качества свиноматок – одни из функциональных признаков, в наибольшей степени влияющих на рентабельность отрасли. При этом проблемы репродукции особей становятся причиной примерно 30% их выбраковки [1].
В последнее время передовые селекционные технологии, основанные на наилучшем линейном несмещенном прогнозе (BLUP), были применены к процессу селекции экономически значимых признаков животных [2, 3]. Понимание генетических параметров данных признаков необходимо для точной оценки племенной ценности и формулирования селекционных индексов в свиноводстве [4, 5]. Между тем генетические параметры специфичны для каждой породы свиней и важно оценивать их отдельно друг от друга, учитывая эти особенности [6].
На сегодняшний день было проведено множество исследований, посвященных оценке генетических параметров продуктивных и репродуктивных признаков свиней, таких как признаки роста [7], характеристики спермы [8] и размер помета [9]. Данные результаты демонстрируют важность генетической оценки экономически значимых признаков для повышения производительности в рассматриваемой отрасли.
Однако большинство свиноматок демонстрируют различную степень физиологической развитости при разных опоросах, что может влиять на репродуктивные способности [10]. Следовательно, получение записей фенотипических характеристик особи при каждом опоросе важно для точной оценки генетических параметров. Кроме того, в аналогичных исследованиях считали разные опоросы одним признаком [2, 11], в то время как другие авторы рассматривали первый и дальнейшие опоросы как отдельные признаки [2, 11–13]. В России аналогичные исследования были проведены на свиньях породы ландрас в 2022 году [14].
Цель исследования – оценить генетические параметры репродуктивных качеств самок свиней крупной белой породы, включая следующие признаки: вес поросят при рождении, вес поросят при отъеме, число живорожденных и мертворожденных поросят, мумифицированного помета. Результаты данной научной работы будут способствовать созданию системы генетической оценки репродуктивных качеств свиноматок и повышению эффективности воспроизводства свиней.
Материалы и методы исследования
Материалом исследования являлись данные первичного учета с программы Herdsman (http://www.herdsman.com) признаков воспроизводства свиноматок крупной белой породы в количестве 500 голов, находящихся в ООО СГЦ «Топ Ген».
В статье встречаются сокращения со следующей расшифровкой: NBA – количество живорожденных поросят; SB – количество мертворожденных поросят; MF – число мумифицированных плодов; LBW – вес поросят при рождении; WW – вес поросят при отъеме.
Описательные статистические параметры выборки представлены в таблице 1.
Таблица 1. Описательная характеристика исследованных признаков в выборке
|
Признак |
Параметр |
Картина распределения |
||
|
M±m |
Min...Max |
Cv, % |
||
|
NBA, гол. |
13,55±0,19 |
0...21 |
25,27 |
|
|
SB, гол. |
0,85±0,07 |
0...9 |
144,39 |
|
|
MF, гол. |
0,29±0,05 |
0...10 |
286,25 |
|
|
LBW, кг |
16,72±0,22 |
0,00...26,70 |
23,63 |
|
|
WW, кг |
93,34±0,79 |
42,00...120,97 |
14,90 |
|
Примечание: CV - коэффициент вариации, также известный как относительное стандартное отклонение, выраженный в %.
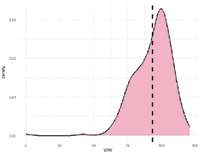
Наблюдаемые картины распределения демонстрируют, что большинство особей характеризовалось нулевыми значениями мертворожденных поросят и мумифицированных плодов. С этим связано существенное смещение распределения, а также высокие значения коэффициента вариации, которые он принимает, исходя из методологии его вычисления. В то же время распределение значений количества живорожденных поросят, массы гнезда при рождении и отъеме было близко к нормальному.
Работа была выполнена в лаборатории функциональной и эволюционной геномики животных ФГБНУ ФИЦ ВИЖ имени Л.К. Эрнста с применением оборудования Центра коллективного пользования научным оборудованием «Биоресурсы и биоинженерия сельскохозяйственных животных» ФГБНУ ФИЦ ВИЖ имени Л.К. Эрнста.
В исследованиях были использованы образцы (фрагмент уха) из коллекции ФГБНУ ФИЦ ВИЖ имени Л.К. Эрнста. Выделение ДНК проводили с помощью наборов для выделения геномной ДНК серии «ДНК-Экстран» (ЗАО «Синтол», Россия) в соответствии с протоколом фирмы-производителя. Концентрацию двухцепочечной ДНК определяли посредством флуориметра Qubit 2.0 (Invitrogen/Life Technologies, США).
Для определения качества ДНК измеряли соотношение OD260/OD280 (cпектрофотометр NanoDrop 8000, Thermo Fisher Scientific, США). Для анализа использовали ДНК с OD260/OD280=1,6–1,8. Кроме того, качество ДНК оценивали посредством гель-электрофореза в 1%-ном агарозном геле.
Полногеномное генотипирование проводили с использованием высокоплотных ДНК-чипов PorcineHD Genotyping BeadChip (платформа GeneSeek Genomic Profiler, Neogene, США), содержащих 66 763 SNP.
Анализ полногеномных ассоциаций осуществлялся с помощью программного обеспечения PLINK 1.90 с учетом популяционной стратификации. Контроль качества и фильтрацию данных генотипирования для каждого SNP и каждого образца выполняли, применяя фильтры call rate по всем исследуемым SNP для индивидуального образца не ниже 90% (-mind 0.10), call rate – для каждого из исследованных SNP по всем генотипированным образцам не ниже 90% (-geno 0.10). Частота встречаемости минорных аллелей (MAF) – ≥0,05, отклонение генотипов по SNP от распределения по Харди-Вайнбергу в совокупности протестированных образцов – ≤0,001 (-hwe 1e-3).
Для поиска генов-кандидатов, локализованных в области идентифицированных SNP, использовали геномный ресурс Sscrofa11.1 (https://www.ncbi.nlm.nih.gov/datasets/genome/GCF_000003025.6/) базы данных NCBI.
Функциональные аннотации и выявление анализа обогащения генов выполняли с привлечением базы данных DAVID (https://david.ncifcrf.gov/summary.jsp) и базы данных Animal QTL (https://www.animalgenome.org/cgi-bin/QTLdb/index).
Результаты и обсуждение
Крупная белая порода свиней является одной из старейших пород (Англия, XIX век), основная ценность которой – многоплодие (как известно, от одной свиноматки можно получить от 14 поросят), а также молочность, выражающаяся в скорости роста подсосного молодняка.
Для геномного тестирования животных в настоящее время используют разработанные тест-системы на маркеры многоплодия – ген ESR (эстрогеновый рецептор), ген FSHB (бета-субъединицы фолликулостимулирующего гормона), ген NCOA1 (ядерный коактиватор А1) и ген EPOR (рецептор эритропоэтина) [15]. Но систему оценки надо улучшать, в том числе расширением применяемых генетических маркеров. Метод GWAS-анализа, представленный в данной статье, является наилучшим на сегодняшний день в вопросе их обнаружения.
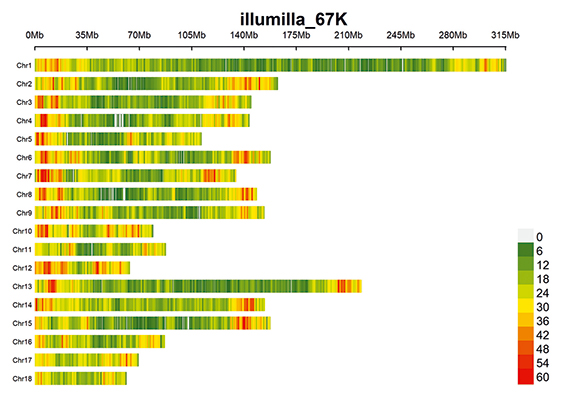
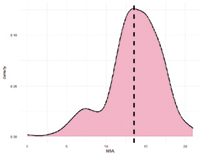
Рис. 1. График плотности SNP по признакам воспроизводства свиноматок крупной белой породы
Выявление обусловленности проявления признака аллельным вариантом гена-кандидата необходимо для создания систем улучшения селекционного отбора по геномной селекции у чистопородных особей. Для визуализации плотности SNP по хромосомам в среде разработки RStudio был использован пакет GWAS rMVP языка R (рис. 1).
После фильтрации данных осталось 51 454 SNP.
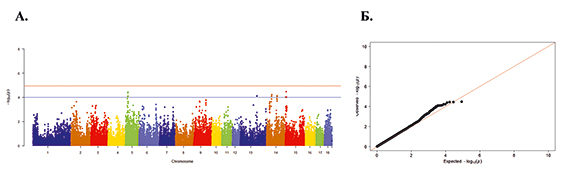
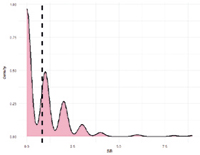
Рис. 2. А. Распределение однонуклеотидных мутаций по хромосомам свиней крупной белой породы в связи с уровнем достоверности (-log10 (P) по вероятностному суггестивному значению (синяя линия; P<0,001) и критерию Бонферрони (красная линия; P<9,72×10-5) для показателя вес поросят при рождении. Б. Квартиль вероятностного распределения ожидаемого и наблюдаемого отклонения от нормального распределения для значений достоверности
Полногеномный анализ по признаку веса поросят при рождении выявил наличие 16 SNP на 5-й, 13-й, 14-й и 15-й хромосомах. Манхэттенский график и Q-Q Plot представлены на рисунке 2.
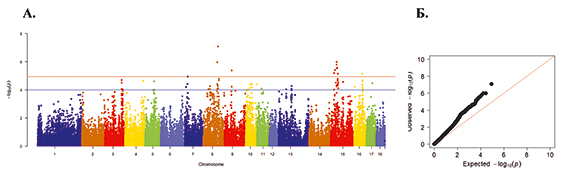
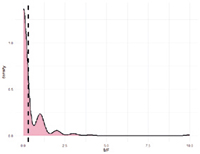
Рис. 3. А. Распределение однонуклеотидных мутаций по хромосомам свиней крупной белой породы в связи с уровнем достоверности (-log10 (P) по вероятностному суггестивному значению (синяя линия; P<0,001) и критерию Бонферрони (красная линия; P<9,72×10-5) для показателя вес поросят при отъеме. Б. Квартиль вероятностного распределения ожидаемого и наблюдаемого отклонения от нормального распределения для значений достоверности
По признаку вес поросят при отъеме обнаружено 79 SNP на 3-й, 4-й, 5-й, 7-й, 8-й, 9-й, 10-й, 11-й, 13-й, 15-й, 16-й и 17-й хромосомах (рис. 3). Наибольшее значение по достоверности у SNP MARC0022286, расположенного на 8-й хромосоме, при Р=8,48х10-8.
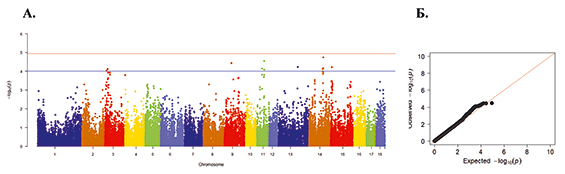
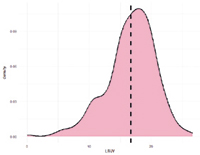
Рис. 4. А. Распределение однонуклеотидных мутаций по хромосомам свиней крупной белой породы в связи с уровнем достоверности (-log10 (P) по вероятностному суггестивному значению (синяя линия; P<0,001) и критерию Бонферрони (красная линия; P<9,72×10-5) для показателя количество живорожденных поросят. Б. Квартиль вероятностного распределения ожидаемого и наблюдаемого отклонения от нормального распределения для значений достоверности
По показателю количества живорожденных поросят выявлено 13 SNP на 3-й, 9-й, 11-й, 13-й, 14-й и 15-й хромосомах, наибольшее по достоверности SNP WU_10.2_3_12326774 на 3-й хромосоме (Р=9,41 х 10-5) (рис. 4).
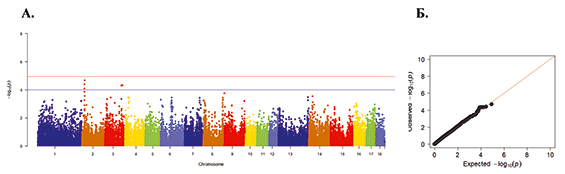
Рис. 5. А. Распределение однонуклеотидных мутаций по хромосомам свиней крупной белой породы в связи с уровнем достоверности (-log10 (P) по вероятностному суггестивному значению (синяя линия; P<0,001) и критерию Бонферрони (красная линия; P<9,72×10-5) для показателя количество мертворожденных поросят. Б. Квартиль вероятностного распределения ожидаемого и наблюдаемого отклонения от нормального распределения для значений достоверности
По показателю количества мертворожденных поросят выявлено 7 SNP на 2-й и 3-й хромосоме (рис. 5 А). График вероятностей представляет собой набор данных под углом 45° – нормальное распределение (рис. 5 Б).
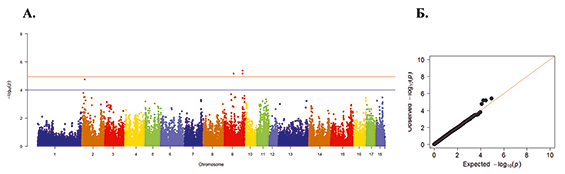
Рис. 6. А. Распределение однонуклеотидных мутаций по хромосомам свиней крупной белой породы в связи с уровнем достоверности (-log10 (P) по вероятностному суггестивному значению (синяя линия; P<0,001) и критерию Бонферрони (красная линия; P<9,72×10-5) для показателя количество мумифицированных плодов. Б. Квартиль вероятностного распределения ожидаемого и наблюдаемого отклонения от нормального распределения для значений достоверности
Одна из патологий, часто встречаемая в свиноводстве, – мумификация плода. Она происходит в результате автолиза и обезвоживания, без мацерации, вызванной размножением бактерий. Процесс мумификации начинается примерно через шесть дней после гибели плода. Причиной данной патологии нередко являются вспышки репродуктивных заболеваний и обычно связаны с вирусными инфекциями (РРСС, парвовирус свиней, вирус болезни Ауески). Также свиноматки могут иметь генетические особенности, делающие их более подверженными мумификации плода.
Генетическое тестирование ускорит процесс выявления склонности животных к мумификации при вынашивании. Так, на рисунке 6 А представлен полногеномный анализ ассоциаций по показателю мумифицированный плод. И по нему установлено достоверных 53 SNP на 4-й, 5-й, 6-й, 9-й, 10-й, 11-й, 14-й, 15-й и 18-й хромосомах. Наибольшее значение достоверности было выявлено у SNP ASGA0062904 и DBNP0002189, находящихся на 14-й хромосоме (Р=6,84х10-8), а наименьшее – у SNP ALGA0060475, ALGA0060479 и ASGA0049456, расположенных на 11-й хромосоме (Р=9,54х10-5).
Всего по пяти исследованным признакам было выявлено 162 SNP, из них 6 SNP были общими для ряда признаков. Дальнейший анализ был направлен на структурную и функциональную аннотацию общих SNP, и по двум признакам из пяти не было обнаружено совпадений.
Данные по общим SNP представлены в таблице 2. Так, для SNP MARC0079305 на 13-й хромосоме были идентифицированы ассоциации с весом при рождении поросят и количеством живорожденных особей (Р=5,94х10-5 и 7,86х10-5). 4 общих SNP обнаружено на 14-й хромосоме: ASGA0062749, ASGA0062912, DBNP0002189 и MARC0010457. Для них были установлены ассоциации с количеством живорожденных поросят, весом поросят при рождении, количеством мумифицированных плодов. Для SNP ASGA0068602 на 15-й хромосоме были выявлены ассоциации с весом при рождении и количеством живорожденных поросят (Р=3,37х10-5 и 6,03х10-5 соответственно).
Таблица 2. Выявленные SNP, общие для воспроизводительных признаков свиноматок крупной белой породы
|
№п/п |
Хр. |
SNP |
Позиция |
Р |
Признак |
|
1 |
13 |
MARC0079305 |
136 916 947 |
5,94E-05 |
NBA |
|
2 |
7,86E-05 |
LBW |
|||
|
3 |
14 |
ASGA0062749 |
39 055 984 |
1,49E-05 |
MF |
|
4 |
8,24E-05 |
LBW |
|||
|
5 |
MARC0010457 |
39 115 601 |
1,49E-05 |
MF |
|
|
6 |
8,17E-05 |
LBW |
|||
|
7 |
ASGA0062912 |
42 013 177 |
1,49E-05 |
MF |
|
|
8 |
8,24E-05 |
LBW |
|||
|
9 |
DBNP0002189 |
42 026 797 |
6,84E-08 |
MF |
|
|
10 |
6,02E-05 |
LBW |
|||
|
11 |
15 |
ASGA0068602 |
6 584 802 |
3,37E-05 |
LBW |
|
12 |
6,03E-05 |
NBA |
Примечание: Хр. – номер хромосомы, на которой расположен SNP, SNP – однонуклеотидный полиморфизм, Р – достоверность SNP.
Расшифровка признаков приведена в разделе «Материалы и методы исследований».
Функциональная аннотация выявила 12 значимых генов, играющих роль в биологической регуляции функций организма (табл. 3). Их можно распределить на следующие группы (GOTERN_BP):
1) клеточные – гены HACD2, RPH3A, SSH1, USP30, DAO;
2) метаболические – гены OAS1 и PTPN11;
3) мышечные и нервные функции – гены MYLK, ADCY5 и CORO1C;
4) процесс формирования костной ткани – гены SELPLG и TMEM119.
Так, в исследованиях X. Deng С et al. на популяции нинсянских и беркширских свиней дифференциально экспрессируемый ген HACD2 может играть жизненно важную роль в жирнокислотном составе липидной ткани и содержании мононенасыщенных и полиненасыщенных жирных кислот в молоке [16]. Также в популяции свиней нинсян молекулярный механизм гена MYLK отвечает за развитие скелетных мышц [17].
Следует отметить ген ADCY5. В разных исследованиях данный ген отвечает за репродуктивные признаки животных и птиц: так, он связан с сезонной течкой у сунских овец [18], яйценоскостью у белых мускусных уток [19] и плодовитостью коров [20], в то время как у человека была обнаружена ассоциация с гестационным периодом беременности [21], а у свиней – со стероидогенезом яичников [22].
Ген RPH3A, согласно литературным данным, участвует в метаболических путях и связывании липидов, а ген OAS1 подавляет репликацию вируса африканской чумы свиней [23]. Ген PTPN11 связывают с репродуктивными процессами у свиней [24].
Таблица 3. Функциональная аннотация выявленных генов-кандидатов.
|
Ген |
SNP |
Функциональная биологическая аннотация по DAVID |
|
HACD2 |
MARC0079305 |
Процесс биосинтеза жирных кислот |
|
MYLK |
Тоническое сокращение гладких мышц |
|
|
ADCY5 |
Баланс контроля нервно-мышечных процессов |
|
|
RPH3A |
ASGA0062749 MARC0010457 |
Внутриклеточный транспорт белков |
|
OAS1 |
Метаболический процесс глюкозы, врожденный иммунный ответ |
|
|
PTPN11 |
Рост и развитие организма, гормональный метаболический процесс |
|
|
SSH1 |
ASGA0062912 DBNP0002189 |
Морфогенез клетки |
|
USP30 |
Слияние митохондрий |
|
|
DAO |
Метаболический процесс D-аминокислоты |
|
|
CORO1C |
Миграция клеток нервного гребня |
|
|
SELPLG |
Процесс формирования костной ткани - окостенение |
|
|
TMEM119 |
процесс формирования костной ткани – оссификация |
Примечание: полужирным шрифтом выделен ген, внутри которого локализован выявленный SNP-маркер.
В базе локусов количественных признаков свиней (pig QTL) из 12 перечисленных генов обнаружен один – ген ADCY5. Ранее он выявлялся с помощью GWAS-анализа у норвежского и датского ландраса как маркер по частоте проявления паховых и мошоночных грыж у свиней (QTL 3047).
Заключение
Поиск новых генетических маркеров, отвечающих за хозяйственно полезные и воспроизводительные параметры животных для их дальнейшего практического применения, является одним из аспектов геномной оценки животных.
В данном исследовании идентификация ассоциаций генов с воспроизводительными качествами свиноматок крупной белой породы осуществлялась с помощью метода полногеномного ассоциативного исследования (GWAS). Выявлено 12 генов, из которых один, а именно ген аденилатциклаза 5 (ADCY5), представляет особый интерес, так как в ряде исследований, проведенных на других видах животных и птиц, было установлено, что он сопряжен с репродуктивными характеристиками.
У свиней этот ген, согласно данным базы локусов количественных признаков, отвечает за предрасположенность к образованию паховых и мошоночных грыж (QTL 3047). В дальнейшем планируется провести исследование частоты генотипа гена ADCY5 и разработать тест-систему с ее апробацией на свиноматках крупной белой породы.
Исследования выполнены за счет проекта РНФ №21-76-10038
Литература
- Onteru S.K. A whole-genome association study for pig reproductive traits/S.K. Onteru, B. Fan, Z.Q. Du, D.J. Garrick, K.J. Stalder, M.F. Rothschild. Anim. Genet., 2012. Vol. 43. P. 18–26. DOI: 10.1111/j.1365-2052.2011.02213.x.
- Zhang T. Hritabilities and genetic and phenotypic correlations of litter uniformity and litter size in Large White sows/T. Zhang, L. Wang, H. Shi, H. Yan, L.-C. Zhang, X. Lui, L. Pu, J. Liang, Y. Zhang, K. Zhao. J. Integr. Agric., 2016. Vol. 15. P. 848–854. DOI: 10.1016/s2095-3119(15)61155-8.
- Gao Y. Short communication: Heritability estimates for susceptibility to Mycobacterium avium ssp. paratuberculosis infection in Chinese Holstein cattle/Y. Gao, J. Cao, S. Zhang, Q. Zhang, D. Sun. J. Dairy Sci., 2018. Vol. 101. P. 7274–7279. DOI: 10.3168/jds.2017-13264.
- Ali B.M. Response to a selection index including environmental costs and risk preferences of producers/B.M. Ali, J.W.M. Bastiaansen, Y. de Mey, A.G.J.M. Lansink. J. Anim. Sci., 2019. Vol. 97. P. 156–171. DOI: 10.1093/jas/sky400.
- Ibanez-Escriche N. Genetic parameters and crossbreeding effects of fat deposition and fatty acid profiles in Iberian pig lines/N. Ibanez-Escriche, E. Magallon, E. Gonzalez, J.F. Tejeda, J.L. Noguera. J. Anim Sci., 2016. Vol. 94. P. 28–37. DOI: 10.2527/jas.2015-9433.
- Darfouroduro K.A. Genetic parameter estimates of growth traits of indigenous pigs in Northern Ghana/K.A. Darfouroduro, A. Naazie, B.K. Ahunu, G.S. Aboagye. Livest. Sci., 2009. Vol. 125. P. 187–191. DOI: 10.1016/j.livsci.2009.04.007.
- Akanno E.C. Meta-analysis of genetic parameter estimates for reproduction, growth and carcass traits of pigs in the tropics/E.C. Akanno, F.S. Schenkel, V.M. Quinton, R.M. Friendship, J.A.B. Robinson/ Livest. Sci., 2013. Vol. 152. P. 101–113. DOI: 10.1016/j.livsci.2012.07.021.
- Li X. Estimation of genetic parameters and season effects for semen traits in three pig breeds of South China/X. Li, B. Jiang, X. Wang, X. Liu, Q. Zhang, Y. Chen. J. Anim. Breed Genet., 2019. Vol. 136. P. 183–189. DOI: 10.1111/jbg.12393.
- Zhang S. Estimation of genetic parameters for reproductive traits in connectedness groups of Duroc, Landrace and Yorkshire pigs in China/S. Zhang, J. Zhang, B.S. Olasege, P. Ma, X. Qiu, H. Gao, C. Wang, Y. Wang, Q. Zhang, H. Yang, Z. Wang, X. Ding, Y. Pan. J. Anim. Breed Genet., 2020. Vol. 137. P. 211–222. DOI: 10.1111/jbg.12431.
- Oh S.H. Estimation of genetic parameters for reproductive traits between first and later parities in pig/S.H. Oh, D.H. Lee, M.T. See. Asian-Australas J. Anim. Sci., 2006. Vol. 19. P. 7–12. DOI: 10.5713/ajas.2006.7.
- Ye J. Genetic parameters for reproductive traits at different parities in Large White pigs/J. Ye, C. Tan, X. Hu, A. Wang, Z. Wu. J. Anim. Sci., 2018. Vol. 96. P. 1215–1220. DOI: 10.1093/jas/sky066.
- Roehe R. Estimation of genetic parameters for litter size in Canadian Yorkshire and Landrace swine with each parity of farrowing treated as a different trait/R. Roehe, B.W. Kenned/ J. Anim. Sci., 1995. Vol. 73. P. 2959–2970. DOI: 10.2527/1995.73102959x.
- Lopez B.I.M. Genetic parameters for litter traits at different parities in purebred Landrace and Yorkshire pigs/B.I.M. Lopez, K. Seo. Anim. Sci. J., 2019. Vol. 90. P. 1497–1502. DOI: 10.1111/asj.13298.
- Белоус А.А. Генетическая архитектура признаков воспроизводства свиней породы ландрас российской репродукции/А.А. Белоус, Волкова В.В., Решетникова А.А., Отраднов П.И., Зиновьева Н.А. Аграрная наука, 2023. №7. С. 31–39. DOI: 10.32634/0869-8155-2023-372-7-31-39.
- Зиновьева Н.А. Молекулярно-генетические методы и их использование в свиноводстве. Достижения науки и техники АПК, 2008. №10. С. 34–36.
- Deng X. Integrative analysis of transcriptomic and lipidomic profiles reveals a differential subcutaneous adipose tissue mechanism among Ningxiang pig and Berkshires, and their offspring/X. Deng, Y. Zhang, G. Song, Y. Fu, Y. Chen, H. Gao, Q. Wang, Z. Jin, Y. Yin, K. Xu. Animals (Basel), 2023. Vol. 13(21). P. 3321. DOI: 10.3390/ani13213321.
- Xu X. Molecular mechanism of MYL4 regulation of skeletal muscle development in pigs/X. Xu, Z. Yu, N. Ai, S. Liufu, X. Liu, B. Chen, X. Li, J. Jiang, Y. Zhang, H. Ma, Y. Yulong. Genes, 2023. Vol. 14. P. 1267. DOI: 10.3390/genes14061267.
- Du X. Comparative transcriptomics reveals the key lncRNA and mRNA of sunite sheep adrenal gland affecting seasonal reproduction/X. Du, X. He, Q. Liu, R. Di, Q. Liu, M. Chu. Front. Vet. Sci., 2022. Vol. 9. P. 816241. DOI: 10.3389/fvets.2022.816241.
- Bello S.F. Hypothalamic and ovarian transcriptome profiling reveals potential candidate genes in low and high egg production of white Muscovy ducks (Cairina moschata)/S.F. Bello, H. Xu, L. Guo, K. Li, M. Zheng, Y. Xu, S. Zhang, E.J. Bekele, A.A. Bahareldin, W. Zhu, D. Zhang, X. Zhang, C. Ji, Q. Nie. Poult. Sci., 2021. Vol. 100. P. 101310. DOI: 10.1016/j.psj.2021.101310.
- Li J. Polymorphic variants of bovine ADCY5 gene identified in GWAS analysis were significantly associated with ovarian morphological related traits/J. Li, C. Shen, K. Zhang, Z. Niu, Z. Liu, S. Zhang, Y. Wang, X. Lan. Gene, 2021. Vol. 766. P. 145158. DOI: 10.1016/j.gene.2020.145158.
- Zhang G. Genetic associations with gestational duration and spontaneous preterm birth/G. Zhang, B. Feenstra, J. Bacelis, X. Liu, L.M. Muglia, J. Juodakis, D.E. Miller, N. Litterman, P.P. Jiang, L. Russell, D.A. Hinds, Y. Hu, M.T. Weirauch, X. Chen, A.R. Chavan, G.P. Wagner, M. Pavličev, M.C. Nnamani, J. Maziarz, M.K. Karjalainen, M. Rämet, V. Sengpiel, F. Geller, H.A. Boyd, A. Palotie, A. Momany, B. Bedell, K. Ryckman, J.M. Huusko, C.R. Forney, L.C. Kottyan, M. Hallman, K. Teramo, E.A. Nohr, G.D. Smith, M. Melbye, B. Jacobsson, L.J. Muglia. N. Engl. J. Med., 2017. Vol. 377. P. 1156–1167. DOI: 10.1056/NEJMoa1612665.
- Sun J. Genome-wide association study on reproductive traits using imputation-based whole-genome sequence data in Yorkshire pigs/J. Sun, J. Xiao, Y. Jiang, Y. Wang, M. Cao, J. Wei, T. Yu, X. Ding, G Yang. Genes (Basel), 2023. Vol. 14(4). P. 861. DOI: 10.3390/genes14040861.
- Sun H. OAS1 suppresses African swine fever virus replication by recruiting TRIM21 to degrade viral major capsid protein/H. Sun, M. Wu, Z. Zhang, Y. Wang, J. Yang, Z. Liu, G. Guan, J. Luo, H. Yin, Q. Niu. J. Virol., 2023. Vol. 97(10). P. e0121723. DOI: 10.1128/jvi.01217-23.
- Zhang W. Population structure and selection signatures underlying domestication inferred from genome-wide copy number variations in Chinese indigenous pigs/W. Zhang, M. Zhou, L. Liu, S. Su, L. Dong, X. Meng, X. Li, C. Wang. Genes (Basel), 2022. Vol. 13(11). P. 2026. DOI: 10.3390/genes13112026.